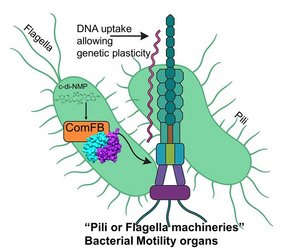
Die Bewegungsorgane (Pili) spielen eine wichtige Rolle bei der DNA-Aufnahme, mit denen verschiedene Bakterien genetische Informationen untereinander austauschen und so die „genomische Plastizität“ ermöglichen. Diese auch als „natürliche Kompetenz“ bezeichnete Fähigkeit beschreibt, dass Bakterienzellen DNA-Moleküle von anderen Bakterien – auch über Artgrenzen hinweg – aufnehmen und in ihr eigenes Genom integrieren können. Die Beweglichkeit der Bakterien und der DNA-Austausch tragen maßgeblich zur Pathogenität, zur Besiedlung von Wirten, zur Bildung von Biofilmen und zur Verbreitung von Antibiotikaresistenzen bei.
In zwei zusammenhängenden Studien entdeckte ein Forschungsteam um Jun.-Prof. Dr. Khaled Selim (Institut für Phototrophe Mikrobiologie der HHU und Exzellenzcluster CMFI der Universität Tübingen) eine neue Familie von Signalproteinen, die im Bakterienreich weit verbreitet sind. Neben der HHU und der Universität Tübingen waren die Universitäten in Gießen und in Freiburg, das Max-Planck-Instituts für Biologie Tübingen, die US-amerikanischen National Institutes of Health (NIH) und die New Jersey Medical School an den Arbeiten beteiligt. Die Forschenden stellten fest, dass die sekundären Botenstoffe c-di-GMP und c-di-AMP zusammen mit dem neu entdeckten ComFB-Rezeptorprotein sowohl die Motilität der Bakterien als auch die DNA-Aufnahme regulieren.
Doktorandin Sherihan Samir, Erstautorin beider Publikationen: „Die verschiedenen Arten von ComFB-Proteinen sind in der Lage, die Motilitäts-/DNA-Aufnahmesignale der Sekundärbotenstoffe c-di-GMP und c-di-AMP zu binden und zu integrieren.“ Gefunden wurden diese Eigenschaften unter anderem in Cyanobakterien, Bacillus subtilis und Vibrio cholerae, dem Erreger der Cholera.
Prof. Selim: „c-di-AMP gehört zu einer kürzlich entdeckten Klasse von ‚zyklischen Dinukleotid-Typ-Sekundärbotenstoffen‘, deren zelluläre Funktionen noch nicht vollständig verstanden waren. In unseren Studien zeigen wir, dass die ComFB-Proteine mit c-di-AMP und zusätzlich mit c-di-GMP binden können und für die Regulierung der genomische Plastizität und der bakteriellen Motilität unerlässlich sind.“
ComFB-Proteine sind im Bakterienreich weit verbreitet. Es ist noch unklar, welche anderen Gruppen von Bakterien – neben den untersuchten – diesen Mechanismus ebenfalls nutzen. „Sollten ihn auch andere pathogene Bakterien mit klinischer Relevanz nutzen, könnte dies den Weg für neue Strategien zur Bekämpfung multiresistenter Bakterien ebnen“, unterstreicht Selim.
Heinrich-Heine-Universität Düsseldorf
Originalpublikationen:
Sherihan Samir, Abdalla A. Elshereef, Vikram Alva, Jeanette Hahn, Frederik Eck, Louisa Celma, Eduardo S. Lopes, Kai Thormann, David Dubnau, Michael Y. Galperin, Khaled A. Selim. ComFB, a new widespread family of c-di-NMP receptor proteins. PNAS 122:38 (2025). https://doi.org/10.1073/pnas.2513041122
Samir, S., Doello, S., Enkerlin, A.M. et al. The second messenger c-di-AMP controls natural competence via ComFB signaling protein. Cell Discov11, 65 (2025). doi.org/10.1038/s41421-025-00816-x