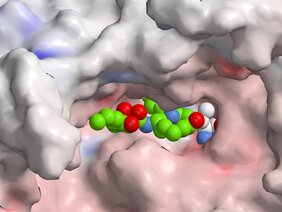
Bevor das Immunystem Eindringlinge wie Bakterien und Viren bekämpfen kann, muss es ihre molekulare Gestalt kennen. Dazu nimmt ein zellulärer Enzymkomplex, das sogenannte Immunoproteasom, den Angreifer auseinander und präsentiert den Immunzellen seine genaue Struktur.
Greift ein überaktives Immunproteasom fälschlicherweise körpereigene Strukturen an, kann es zu Immunerkrankungen kommen. Um sie zu regulieren, suchen Forschende seit langem nach Hemmstoffen gegen das Immunoproteasom. Dabei muss jedoch sichergestellt sein, dass die anderen Proteasom-Varianten der Zelle – zum Beispiel solche, die für das zelluläre Recycling und die Abfallentsorgung notwendig sind – nicht ebenso blockiert werden. Selektivität der Wirkstoffe ist hier höchstes Gebot, um Nebenwirkungen zu vermeiden.
Die Arbeitsgruppe von Prof. Helge Bode am Max-Planck-Institut für terrestrische Mikrobiologie in Marburg forscht seit Jahren daran, Enzymkomplexe am Reißbrett zu designen und mithilfe der synthetischen Biologie neue Naturstoffe zu erzeugen.
Bisherige mögliche Kandidaten für Medikamente gegen Immunerkrankungen, aber auch Antibiotika oder solche gegen Krebs, stammen nicht nur aus der Stoffgruppe der Peptide (= Eiweiße), sondern auch aus der Gruppe der langkettigen Fettsäuren, sogenannter Polyketide. Während Peptide meist durch nicht-ribosomale Peptidsynthetasen (NRPS) produziert werden, entstehen Polyketide durch Polyketidsynthasen (PKS).
In Zusammenarbeit mit Prof. Dr. Michael Groll (TU München) und Prof. Dr. Markus Kaiser (Universität Duisburg-Essen) gelang es dem Team, ein Hybrid aus Peptid und Polyketid zu entwickeln und in wenigen Schritten zusammenzubauen. „Die von uns entwickelte XUT Technologie nutzt Schnittstellen, die in sogenannten Thiolierungs- (T) Domänen vorkommen. Da es diese T-Domänen sowohl in NRPS und PKS gibt, kann man auch beide Sorten von Enzymen, Peptide und Polyketide, miteinander fusionieren“, erklärt Leonard Präve, Erstautor der Studie, die online in der Fachzeitschrift Chem erschienen ist.
Solche Hybride aus NRPS und PKS stellt die Natur auch selbst her. Eine bestimmte Substanzklasse, die Syrbactine, finden sich z.B. bei Bakterien, die Pflanzen oder auch Insekten schädigen. Indem Syrbactine das Proteasom in diesen höheren Organismen hemmen, stirbt die Zelle durch „Verstopfung“ ihrer Abfallentsorgung. Da eben diese Wirkung im Falle von Tumorzellen wünschenswert ist, gelten Syrbactine als Kandidaten für Krebsmedikamente. Zwar gibt es bereits Medikamente, die auf der Hemmung des Proteasoms beruhen, doch gab es jedoch bislang keinen biotechnologischen Zugang zu einem spezifischen und damit nebenwirkungsarmen Wirkstoff gegen das Immunoproteasom aus der Klasse der Syrbactine.
„Mit unserer Technik konnten wir die Syrbactine in mehreren Schritten ganz rational so verändern, dass erstmals ein neuartiger, selektiverer Hemmstoff gegen das humane Immunoproteasom entsteht“, ergänzt Helge Bode. Zwar ist der erzeugte Wirkstoff noch nicht selektiv genug, zeigt aber schon die Richtung auf, wie weitere Varianten nun optimiert werden können, um Nebenwirkungen zu reduzieren. Diese sollen in Zukunft am Reißbrett und im hohen Durchsatz erzeugt werden, um so die besten Varianten für die spezifische Anwendung aussuchen zu können.
Max-Planck-Institut für terrestrische Mikrobiologie
Originalpublikation:
Präve, L.; Kuttenlochner, W.; Tabak, W. A. W.; Langer, C.; Kaiser, M.; Groll, M.; Bode, H. B.: Bioengineering of Syrbactin Megasynthetases for Immunoproteasome Inhibitor Production, Chem 2024, https://doi.org/10.1016/j.chempr.2024.07.013