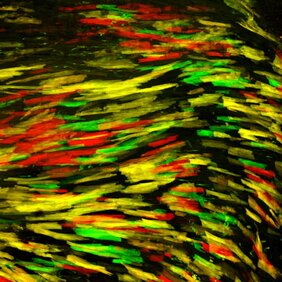
Individuen, die ein „genetisches Mosaik“ bilden, also Zellen mit unterschiedlichen Genotypen enthalten, kommen bei mehrzelligen Organismen natürlich vor. Beim Menschen ist die Entwicklung von Krebs – wobei eine Zelle eine Mutation erwirbt, die es ihr erlaubt, sich zu vermehren, während andere Zellen dies nicht tun – ein Paradebeispiel für ein genetisches Mosaik. Umgekehrt können genetische Mosaike aber auch genutzt werden, um die Entstehung von Krankheiten zu untersuchen und zu verstehen.
Eine häufige Laune der Natur, nützlich zum Verständnis von Genen
Ein experimenteller genetischer Mosaik-Ansatz ist MADM (Mosaic Analysis with Double Makers). Bei dieser Methode werden Gene in einzelnen Zellen mutiert und die mutierten Zellen gleichzeitig in fluoreszierenden Farben markiert. „MADM ist eine Markierungstechnologie, mit der wir im Prinzip Zellen markieren können, in denen jedes für uns interessante Gen mutiert sein kann, und zwar in jedem Organ", erklärt Erstautor Simon Hippenmeyer. Da das Gen in einer einzelnen Zelle verändert wird, während die übrigen Zellen „normal" bleiben, können die WissenschaftlerInnen verfolgen, was mit der mutierten Zelle passiert und so Einblicke in die Rolle und Funktion des mutierten Gens gewinnen. Dieser Ansatz ist besonders wertvoll bei essenziellen Genen: Die Mutation eines essenziellen Gens in allen Zellen eines Organismus würde die Gesundheit und Lebensfähigkeit des Organismus beeinträchtigen. Wird das Gen jedoch in nur wenigen ausgewählten Zellen mutiert, ist der Organismus selbst nicht betroffen. Trotzdem können die WissenschaftlerInnen auf Einzelzell-Ebene verfolgen, was mit den wenigen mutierten Zellen geschieht und ihre Morphologie, Entwicklung und Funktion beobachten.
Bisher konnten mit der MADM-Technik nur etwa 25 Prozent der Gene im Mausgenom mutiert und verfolgt werden, da die MADM-Technik auf drei der Mäuse-Chromosome beschränkt war. Jetzt haben Hippenmeyer und seine Gruppe am IST Austria diese Möglichkeit dramatisch erweitert. Die Gruppe hat die für die MADM-Technik erforderliche „MADM-Markierungskassette" erfolgreich auf allen Mäusechromosomen platziert (außer auf den Geschlechtschromosomen). Nun können mehr als 96% der Gene mit MADM auf Einzelzellebene mutiert und verfolgt werden. „Wir können jetzt fast jedes Gen der Maus einfach manipulieren und jedes Gen einer hochauflösenden, phänotypischen Genmosaikanalyse unterziehen", erklärt Hippenmeyer.
Neue Wege für die Krebsforschung
Hippenmeyer erwartet, dass diese Ressource einen Schub für die Erforschung von Krankheiten und allgemeinen Entwicklungsmechanismen bedeutet. „Jetzt können wir Gene untersuchen, die mit Krankheiten assoziiert sind, die durch eine einzige mutierte Zelle entstehen. Krebs ist das Paradebeispiel dafür. Mit unserer Ressource kann systematisch jedes einzelne bekannte Tumorsuppressor-Gen und seine Rolle bei der Krebsentstehung und -evolution untersucht werden, auch in Kombination mit anderen Mutationen." In den letzten Jahren haben ForscherInnen MADM für mehrere Krebsstudien eingesetzt, unter anderem für das Screening nach Wirkstoff-Zielen. „Unsere MADM-Bibliothek ist nicht nur eine Möglichkeit, den Krankheitsverlauf zu analysieren, sondern bietet auch eine Plattform für die Entdeckung von Medikamenten und Wirkstoffzielen", fügt Hippenmeyer hinzu. „Dies ist nicht auf Krebs beschränkt: MADM kann auch verwendet werden, um Krankheiten in vielen Kontexten zu untersuchen und zu verstehen. Darunter fallen auch Störungen der neuronalen Entwicklung sowie andere Gehirnerkrankungen, die das Hauptinteresse der Hippenmeyer-Gruppe sind."
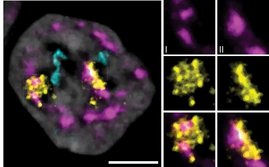
In ihrer Studie nutzten Hippenmeyer und seine Gruppe die neue Ressource, um das Anwendungsspektrum von MADM zu erweitern und ein faszinierendes Problem in der Biologie zu beleuchten. Sie fanden Hinweise darauf, dass die Chromosomensegregation während der asymmetrischen Zellteilung einem nicht zufälligen Muster folgt. „Unsere Ergebnisse zeigen zum ersten Mal in vivo, dass die Art und Weise, wie sich die elterlichen Chromosomen während der Stammzellteilung segregieren, das zelluläre Schicksal der entstehenden Tochterzellen beeinflussen könnte. In einem breiteren Kontext sind diese Erkenntnisse relevant für unser allgemeines Verständnis der Stammzellbiologie und vielleicht auch für die Mechanismen der Krebsentstehung."
In Zukunft wird der Neurowissenschaftler Hippenmeyer die erweiterten Möglichkeiten von MADM nutzen, um das Verhalten von Stammzellen während der Gehirnentwicklung zu untersuchen. Ein Hauptaugenmerk liegt dabei auf den Mechanismen, die dafür sorgen, dass sich Gehirne in der richtigen Größe entwickeln. Beim Menschen sind Störungen der Gehirngröße, wie Mikro- und Makroenzephalie, mit Epilepsie und geistiger Behinderung verbunden. „Wir können nun fragen, was in einer Stammzelle schief läuft, so dass sich das Gehirn zu groß oder zu klein entwickelt. Wir gehen davon aus, dass unsere Ergebnisse in Zukunft auch eine Grundlage für die stammzellbasierte Reparatur und Regeneration des Gehirns bilden können".
IST Austria
Originalpublikation:
Contreras, Amberg, Davaatseren, Hansen, Sonntag, Andersen, Bernthaler, Streicher, Heger, Johnson, Schwarz, Luo, Rülicke & Hippenmeyer. 2021. A Genome-wide Library of MADM Mice for Single-Cell Genetic Mosaic Analysis. Cell Reports. DOI: 10.1016/j.celrep.2021.109274