Zelllinien werden aus Krebszellen gewonnen und sind in der Grundlagenforschung als Modellsysteme etabliert. In der Corona-Forschung werden diese Modellsysteme unter anderem genutzt, um das Eindringen des Virus in die Zelle und die anschließende Vermehrung und Freisetzung der Viruspartikel insbesondere auch unter Einwirkung von Medikamenten zu untersuchen. Bislang hat sich die Forschung auf drei Zelllinien fokussiert, von denen eine aus dem Affen stammt. „Die Auswahl einer geeigneten Zelllinie ist Voraussetzung für die Grundlagenforschung. Denn Ergebnisse, die in tierischen Zellen erhalten wurden, können nur bedingt auf menschliche Zellen und damit auf den Menschen übertragen werden“, betont Zellbiologe Dr. Cord Uphoff vom Leibniz-Institut DSMZ. Zusammen mit seinen Kolleginnen und Kollegen hat er mehr als 300 menschliche Krebszelllinien auf ihre Tauglichkeit als Modellsystem untersucht.
ACE2-Rezeptor ist nicht ausreichend für ein Modellsystem
In ihren Experimenten konnten die Forschenden an der DSMZ und dem HZI zeigen, dass das Vorhandensein der Oberflächenproteine ACE2 und TMPRSS2 nicht allein ausschlaggebend dafür ist, dass SARS-CoV-2 in die Zelle eindringen kann. Veränderungen in der Zusammensetzung der Oberflächenproteine, bedingt etwa durch den Austausch von Aminosäuren, können die Aufnahme des Coronavirus in die Zelle erheblich beeinflussen. Auch andere Parameter wie verfügbare Ressourcen innerhalb der Zelle, ihre intrinsische Immunität oder anstehende Apoptose müssen berücksichtigt werden.
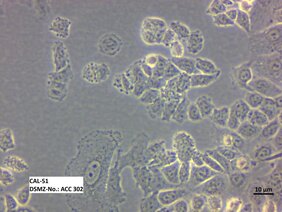
CL-14, CL-40 und CAL-51 für die Corona-Forschung
Nach bioinformatischen Analysen wählten die Forschenden 29 Zelllinien aus, die in Experimenten im Labor der biologischen Schutzstufe 3 des HZI von Dr. Ulfert Rand dem SARS-CoV-2 ausgesetzt wurden. „Frei zugängliche Datenbanken, wie in diesem Fall die Cancer Cell Line Encyclopedia, ermöglichen diese Art von Screening. Die Vorauswahl spart dem wissenschaftlichen Personal viel Zeit und Ressourcen. Sie können direkt mit den Laboruntersuchungen beginnen.“, sagt Bioinformatikerin Dr. Claudia Pommerenke vom Leibniz-Institut DSMZ. Die Ergebnisse der Laborversuche waren jedoch unerwartet: der größte Teil der Zelllinien, die aufgrund vorhandener ACE2- und TMPRSS2-Rezeptoren vom Coronavirus hätten befallen werden sollen, reagierten gar nicht auf das Virus, während elf Zelllinien lediglich geringe Mengen infektiöses Virus bildeten. Nur bei drei Zelllinien konnte eine hohe Aufnahme, Vermehrung und Freisetzung von Corona-Viruspartikeln festgestellt werden. Dabei handelt es sich um die Zelllinien CL-14 und CL-40 (menschliche Darmkrebszellen) und CAL-51 (menschliche Brustkrebszellen). „Die drei Zelllinien eignen sich aufgrund ihrer Eigenschaften hervorragend als Modellsystem in der Corona-Forschung. Die Zellen sind von uns bestens charakterisiert und befinden sich in der Sammlung des Leibniz-Instituts DSMZ. Sie können von Forschenden weltweit für Experimente bestellt werden.“, fasst Cord Uphoff zusammen.
Mehr als 860 Zelllinien in der DSMZ Sammlung Menschlicher und Tierischer Zellkulturen
Die Sammlung des Leibniz-Instituts DSMZ umfasst mehr als 860 Zelllinien menschlichen und tierischen Ursprungs, darunter auch das LL-100 panel. Dies umfasst 100 verschiedene Krebszelllinien aus 22 Tumor-Entitäten von menschlichen Leukämien und Lymphomen.
Leibniz-Institut DSMZ-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH
Originalpublikation:
Pommerenke C., Rand U., Uphoff C., Nagel S., Zaborski M., Hauer V., Kaufmann M., Meyer C., Denkmann S., Riese P., Eschke K., Kim Y., Safranko Z.M., Kurolt I.C., Markotic A., Cicin-Sain L., Steenpass L. (2021) Identification of cell lines CL-14, CL-40 and CAL-51 as suitable models for SARS-CoV-2 infection studies. PLoS One. 2021 Aug 2;16(8):e0255622. doi: 10.1371/journal.pone.0255622.