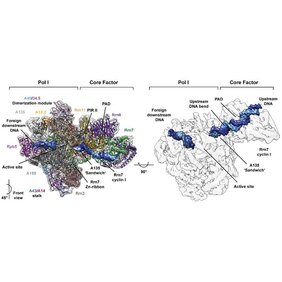
Die Struktur zeigt, wie eine native DNA-Sequenz von Proteinen des ‘Core Factor‘ erkannt wird. Durch die Erkennung kann Pol I hochspezifisch rekrutiert werden und direkt mit der Herstellung von rRNA begonnen werden. Um diese Pol I Aktivität zu ermöglichen, muss doppelsträngige DNA zuerst in ihre Einzelstränge getrennt (d. h. geschmolzen) werden. Die Regensburger Wissenschaftler fanden heraus, dass sich die Mechanismen der Rekrutierung von Pol I und des Schmelzens der DNA grundlegend von verwandten Systemen unterscheiden. Biochemische Analysen der DNA-Bindungsstärke des Core Factors und der spezifischen Aktivität der Pol I untermauern diese Vermutung. Des Weiteren haben sich strukturelle Elemente im Core Factor entwickelt, die anscheinend zur erfolgreichen DNA-Schmelzung beitragen. Folglich konnten die Biochemiker zeigen, dass eine Entfernung dieser Elemente im Core Factor dazu führt, dass Pol I ihre Aufgabe nicht mehr ausführen kann. Abschließend hat eine vergleichende Strukturanalyse ergeben, dass es während der Rekrutierung zur Kontraktion des Enzyms Pol I kommt. Diese Kontraktion drückt doppelsträngige DNA praktisch zusammen und unterstützt dadurch die Trennung der beiden Einzelstränge voneinander. Ein ähnlicher Mechanismus bleibt verwandten Enzymen aufgrund ihrer strukturellen Eigenheiten verwehrt. Die Forscher gehen deshalb davon aus, dass sich Pol I im Laufe der Evolution spezifisch an ihre Aufgabe angepasst hat und daher äußerst effizient agieren kann.
Dank dieser Ergebnisse können die Wissenschaftler nun neue mechanistische Fragestellungen bearbeiten. Beispielsweise: Wie wird festgelegt, an welche DNA-Sequenzen Core Factor und damit Pol I rekrutiert werden? Und: Was ist die Rolle von regulatorischen Proteinen und deren Modifikationen? Zusätzlich zur Grundlagenforschung erfolgen Bemühungen, Pol I künstlich gezielt zu hemmen. Da vor allem schnell wachsende Zellen, wie z. B. Krebszellen, eine hohe Pol I Aktivität aufweisen, wird eine chemotherapeutische Nutzung solcher Strategien derzeit getestet. Die Analysen der Biochemiker können diese Bestrebungen nun mit Erkenntnissen auf struktureller Ebene unterstützen.
Universität Regensburg
Originalpublikation:
Pilsl M and Engel C: Structural basis of RNA polymerase I pre-initiation complex formation and promoter melting. Nature Communications 2020. DOI: 10.1038/s41467-020-15052-y