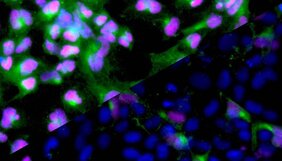
Unter dem Mikroskop erscheint unser Erbgut wie ein wild in den Zellkern gestopftes Wollknäuel. Und doch herrscht hier kein Chaos, denn fast jedes Knötchen und jede Schlaufe im Erbgutfaden hat auch eine Funktion. In den „Loop-Domänen“ genannten DNA-Schlaufen versammeln sich meist Gene zusammen mit Sequenzabschnitten, die die Genaktivität präzise steuern.
Wie ein internationales Forschungsteam nun herausfand, könnten die Loop-Domänen offenbar dazu dienen, Gene zu isolieren und so vor Störungen durch andere DNA-Regionen zu schützen. Denn insbesondere Gene, die während der Embryonalentwicklung besondere wichtige Aufgaben haben, beanspruchen im menschlichen Erbgut eine Schlaufe nur für sich und ihre regulatorischen Sequenzen. Lösten die Forschenden zum Beispiel die Schlaufe um das Entwicklungsgen SOX17 auf, war die Entwicklung von embryonalen Zellen beeinträchtigt.
Diese Ergebnisse stellte das Forschungsteam vom Max-Planck-Institut für molekulare Genetik (MPIMG) in Berlin, dem Dana-Farber Cancer Institute in Boston, USA, der Universität Harvard in Cambridge, USA und der Universität Peking, China nun im Fachblatt Nature Communications vor.
„Uns interessiert, welche Bedeutung die dreidimensionale Genomstruktur in humanen Stammzellen hat“, sagen Alexander Meissner vom MPIMG und Franziska Michor vom Dana-Farber Cancer Institute und der Universität Harvard, Hauptautoren der Studie. „Bislang hatte noch niemand systematisch den Zusammenhang zwischen Genfunktion und Anzahl der Gene innerhalb von Loop-Domänen beim Menschen hergestellt.“
Die DNA-Schlaufen bilden dreidimensionale Funktionseinheiten, die auch unter dem Namen topologisch assoziierte Domänen (TAD) firmieren. Zusammengehalten werden sie unter anderem durch die Proteine CTCF und Cohesin, die das Erbgut wie ein Kordelstopper an spezifischen Sequenzabschnitten zusammenziehen und den Bereich dazwischen in eine Schlaufenform bringen.
In einer TAD-Schlaufe befinden sich meist ein oder mehrere Gene und deren regulatorische Elemente. Diese interagieren intensiv miteinander, während es über die Grenzen der Domäne hinaus kaum Wechselwirkungen gibt. Knapp die Hälfte aller proteinbildender Gene befinden in solchen Schlaufen – allein oder zusammen mit weiteren Genen. Die seltenen Störungen dieser Strukturen an Orten mit entwicklungsrelevanten Genen ziehen Entwicklungsstörungen und Fehlbildungen nach sich.
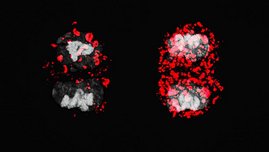
Die Forscherinnen und Forscher nutzten zunächst embryonale Stammzellen des Menschen für ihre Untersuchungen. Durch das Verfahren „Hi-C“ konnten sie messen, welche Bereiche des Erbguts sich durch die Faltung bevorzugt berühren. Über die Kontaktpunkte errechneten sie dann die 3D-Struktur der DNA, also wie der DNA-Strang im Raum angeordnet ist.
„Es hat uns sehr überrascht, dass sich in fast 40 Prozent der Loop-Domänen nur ein einziges Gen befindet,“ sagt Hua-Jun Wu von der Universität Peking, einer der Erstautoren der Studie. Ein Fünftel der Erbgutschlaufen ist dagegen leer und enthält gar keine Gene, etwa zwei Fünftel sind mit mehreren Genen bestückt.
In den genetischen Séparées fanden die Forscherinnen und Forscher zwar Gene aus allen Funktionsbereichen der Zelle. „Besonders Gene mit einer essentiellen Funktion für die Embryonalentwicklung scheinen sich einzeln in Loop-Domänen zurückzuziehen. Im Vergleich zu allen anderen Genen sind sie dort zu 50 Prozent überrepräsentiert“, sagt der Forscher.
Über einen Zeitraum von bis zu sechs Wochen der Embryonalentwicklung betrachtet, blieben die Domänen mit einem einzigen Gen stabil. Die Entwicklungsgene sowie die zugehörigen Regulatoren blieben also im frühen Embryo dauerhaft unter sich.
„Die Isolation der wichtigsten Gene in klar definierte Bereiche, die vor äußeren Einflüssen geschützt sind, ergibt Sinn“, sagt Wu. „Entwicklungsgene unterliegen ohnehin einer besonders strengen Kontrolle – die kleinste Änderung in der Genaktivität kann weitreichende Konsequenzen haben, also zum Beispiel die Regulation anderer Gene beeinflussen und damit den gesamten empfindlichen Prozess der Embryonalentwicklung stören.“
Tatsächlich ist das Prinzip der genetischen Isolation im Reich der Säugetiere weit verbreitet und hat sich im Laufe der Evolution bewährt, stellten die Forschenden fest. Sie verglichen die Sequenzdaten von 45 Tierarten und fanden, dass sich die Grenzen von Domänen mit nur einem Gen im Laufe der Evolution von Säugern nicht oder kaum verändert haben.
Was passiert aber, wenn ein isoliertes Gen sein Privileg verliert? Dieser Frage gingen die Forschenden an induzierten pluripotenten Stammzellen (iPS-Zellen) nach. Die Wissenschaftlerinnen und Wissenschaftler entfernten gezielt die Loop-Struktur um das Gen SOX17. Das Entwicklungsgen ist wichtig für die Ausbildung der inneren Zellschichten des Embryos, das Endoderm, und damit für die Entwicklung von Organen wie Leber, Bauchspeicheldrüse und Darm. Zudem ist es für seine besonders strenge Isolation bekannt.
Die Forschenden schnitten den Bereich aus dem Erbgut, mit dessen Hilfe sich der DNA-Strang wie eine Schlinge zusammen zieht, und beobachteten, wie sich die Genaktivität veränderte. „Das SOX17-Gen liegt in einem klassischen Enhancer-Loop. Verliert es den Kontakt zu seinem eigenen Regulator, dem Enhancer, kann es nicht mehr aktiviert werden“, sagt Alexandro Landshammer, ebenfalls Erstautor der Arbeit. „Die Regulatoren anderer Gene können SOX17 nicht retten, und so haben wir in unseren Experimenten nur noch sehr wenige Zellen gesehen, in denen das Gen noch eingeschaltet war.“
Aber nicht nur die Aktivität von SOX17 blieb aus, auch weitere Gene, die für die Ausbildung der Endoderm-Zellen wichtig sind, blieben stumm. Denn SOX17 aktiviert seinerseits weitere Entwicklungsprogramme und damit neue Gene.
Als Gegenbeispiel wählten die Wissenschaftlerinnen und Wissenschaftler das Gen NANOG, das sich eine Erbgutschlaufe mit mehreren anderen Genen teilt. Hier hatte das Auflösen der Loop-Domäne keinen messbaren Effekt – um isolierte Gene scheinen die Domänen also eine besonders wichtige Rolle zu spielen.
„Wir glauben, dass Loop-Domänen ein Sicherheitsmerkmal des Genoms sein könnten. Die isolierten Gene werden vor den Einflüssen der benachbarten Sequenzabschnitte abgeschirmt“, sagt Meissner. „Denn die Embryonalentwicklung folgt einem festen Fahrplan, es müssen Gene in spezifischen Geweben für genau definierte Zeitspannen aktiviert werden. Kleinste Abweichungen können verheerende Folgen haben.“
„Verschiedene Gene sind möglicherweise nicht gleich empfindlich gegenüber Störungen“, sagt Meissner. Eine Fehlregulation von somatischen Genen führe womöglich zu Fehlbildungen oder Tumoren, seien aber nicht unmittelbar tödlich für den Organismus. Um diese These zu belegen, müsse man die Loop-Domänen weiter systematisch untersuchen, sagt der Forscher.
Nichtsdestotrotz sehen die Wissenschaftlerinnen und Wissenschaftler Chancen für Therapieformen, die die 3D-Struktur der DNA beeinflussen – einzelnen Forschungsteams ist dies bereits gelungen. „Die spannenden Entwicklungen in der Forschung lassen hoffen, dass Mutationen in Domänengrenzen einmal als diagnostische Marker oder Zielstrukturen für Medikamente dienen könnten“, sagt Michor. „Unser Studie hilft vielleicht, den Fokus auf die essentiellen und streng regulierten Gene der frühen Entwicklung des Embryos zu lenken.“
Max-Planck-Institut für molekulare Genetik
Originalpublikation:
Wu HJ, Landshammer A, Stamenova EK, Bolondi A, Kretzmer H, Meissner A, Michor F. Topological isolation of developmental regulators in mammalian genomes. Nat Commun. 2021 Aug 12;12(1):4897. doi: 10.1038/s41467-021-24951-7