Prof. Dr. Matthias Meier vom Biotechnologisch-Biomedizinischen Zentrum der Universität Leipzig und seine Arbeitsgruppe haben in Zusammenarbeit mit dem Helmholtz Zentrum München eine neue, effektive und vergleichsweise kostengünstige Methode entwickelt, um seltene Zelltypen, Zellkommunikationsarten oder Krankheitsmuster im Gewebe sichtbar zu machen. Ihre Erkenntnisse haben die Forschenden gerade in dem renommierten Fachjournal „Nature Communications“ veröffentlicht.
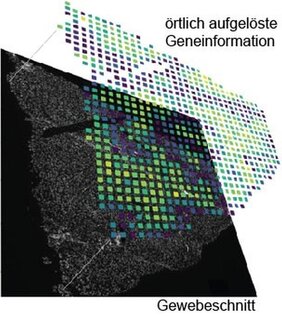
Für alle Einzelzell-Analyse-Methoden müssen Zellen aus dem Gewebeverbund gelöst werden, wobei die räumliche Information der Zelltypen und damit die Information der zellulären Umgebung, zelluläre Kommunikationswege oder Funktion verloren gehen. Um räumlich aufgelöste Einzelzell-Informationen zu gewinnen, müssen Bildgebungs- und Sequenzierungstechniken in Kombination arbeiten. Mehrere Ansätze wurden in den vergangenen Jahren entwickelt, um die Zusammenführung von Bild- und Sequenzierdaten zu vereinigen.
Dabei wurden je nach Forschungsfrage verschiedene Parameter wie räumliche Auflösung, Nachweisgrenze, Zugänglichkeit der Ribonukleinsäuren und Kosten gegeneinander abgewogen. Ein früheres Analyseverfahren nutzte die Idee, die örtliche Information mittels eines Strichcodes basierend auf der Abfolge von DNA-Basen an Ribonukleinsäuren zu hängen. Nach Extraktion aller Ribonukleinsäuren und anschließendem massenhaften Sequenzieren können die Strichcodes dazu verwendet werden, um ein künstliches Bild zu generieren.
Hier setzt die Arbeit von Johannes Wirth an. Als Doktorand im Labor von Matthias Meier hat der Forscher vom Helmholtz Zentrum München einen fortschrittlichen Arbeitsablauf entwickelt, der es ermöglicht, örtlich aufgelöste genomische Daten gepaart mit hochwertigen Mikroskopiebildern aufzunehmen. Dadurch wird es möglich, seltene Zelltypen, Zellkommunikationsarten oder Krankheitsmuster im Gewebe sichtbar zu machen.
Im Zentrum stand die Entwicklung eines neuen mikrofluidischen Chips, der es ermöglicht, Ribonukleinsäureketten in großflächigen Gewebeabschnitten kostengünstig zu analysieren. „Im Vergleich zur ursprünglichen Methode konnte im neuen Ansatz die Anzahl der Bildinformationen pro Bildpunkt um das Sechs- beziehungsweise Zwölffache erhöht werden. Umgekehrt bedeutet dies, dass wir in der Lage sind, etwa 5.000 Gene pro Bildpunkt aufzulösen, so dass seltene Zelltypen in der Niere oder Leber sichtbar gemacht werden können“, erklärt Wirth. Im Vergleich dazu könne ein Standard-HD-Bildschirm pro Bildpunkt nur die drei Primärfarben mit 256 verschiedenen Helligkeitsstufen darstellen.
Zusätzlich zu den technischen Fortschritten stellte das Team auch eine Open-Source-Analysepipeline zur Verfügung, um die Methode leicht zugänglich zu machen. Da das Verfahren für eine Vielzahl von Geweben geeignet ist, wird es Studien zu komplexen Krankheiten und Multiorgan(dys-)funktionen erleichtern. „Das von uns entwickelte Verfahren, bei dem Bildgebungs- und Sequenzierungstechniken in Kombination arbeiten, war bis vor kurzer Zeit noch eine Vision. Es hat unser Verständnis der zellulären Heterogenität revolutioniert und uns neue Zelltypen in allen Organismen finden lassen“, sagt Prof. Meier. Durch die Entwicklung von Einzelzell-Sequenziermethoden sei es heute möglich, zelluläre Entwicklungswege oder Krankheitsverläufe in Tiefe besser zu verstehen.
Universität Leipzig
Originalpublikation:
Wirth, J., Huber, N., Yin, K. et al. Spatial transcriptomics using multiplexed deterministic barcoding in tissue. Nat Commun14, 1523 (2023). https://doi.org/10.1038/s41467-023-37111-w