In jeder einzelnen Zelle unseres Körpers liegt unsere DNA, die Bausteine für unsere Entwicklung und unser Wachstum, verpackt mit Proteinen in bestimmten Strukturen - den Chromosomen. Vollständige Chromosomensätze bilden zusammen das Genom, das die genetische Information eines Organismus enthält.
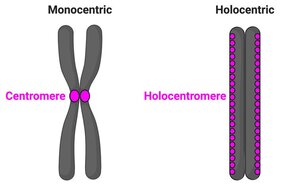
Bei den meisten Organismen, so auch bei uns, erscheinen die Chromosomen als X-förmige Strukturen, wenn sie in ihrem kondensierten, duplizierten Zustand zur Vorbereitung der Zellteilung vorliegen. Diese Strukturen gehören zu den symbolträchtigsten in der gesamten Wissenschaft. Die X-Form ist auf eine verengte Region zurückzuführen, ähnlich wie ein Gürtel der sich um die Chromosomen legt, das sogenannte Zentromer. Es dient dazu, die Schwesterchromatiden zu verbinden, d. h. die identischen Kopien, die bei der DNA-Verdoppelung eines Chromosoms entstehen, aber auch um diese während der Zellteilung zu transportieren. Die meisten untersuchten Organismen sind "monozentrisch", d. h. die Zentromere sind auf eine einzige Region auf jedem Chromosom beschränkt. Einige tierische und pflanzliche Organismen weisen jedoch eine ganz andere Organisation von Zentromeren auf: Anstelle einer einzigen Einschnürung wie bei den klassischen X-förmigen Chromosomen weisen die Chromosomen dieser Organismen mehrere Zentromere auf, die in einer Linie entlang Schwesterchromatiden angeordnet sind. Diesen Chromosomen fehlt also eine primäre Einschnürung und die X-Form. Arten mit solchen Chromosomen werden als "holozentrisch" bezeichnet, vom altgriechischen Wort hólos, das "ganz" bedeutet.
Forschende unter der Leitung von André Marques vom Max-Planck-Institut für Pflanzenzüchtungsforschung konnten nun die bemerkenswerten Auswirkungen dieser nicht-klassischen Form der Chromosomenorganisation auf die Genomarchitektur und die Evolution zeigen.
Um herauszufinden, wie sich eine holozentrische Chromosomenorganisation auf das Genom auswirkt, entschlüsselten Marques und sein Team mit Hilfe einer hochpräzisen DNA-Sequenzierungstechnologie die Genome von drei eng verwandten holozentrischen Schnabelrieden, die zur Familie der Sauergrasgewächse gehören, die weltweit vorkommen und oft die ersten Eroberer neuer Lebensräume sind. Als Referenz entschlüsselte das Team das Genom des am engsten verwandten monozentrischen Verwandten. Durch den Vergleich der holozentrischen Schnabelriede mit ihren monozentrischen Verwandten konnten die Autoren anhand der beobachteten Unterschiede die Auswirkungen der Holozentrizität analysieren.
Ihre Analysen zeigen auffällige Unterschiede in der Genomorganisation und Chromosomenevolution in holozentrischen Organismen. Die Zentromerfunktion in holozentrischen Arten liegt verteilt über Hunderte von kleinen Zentromerdomänen entlang der Chromosomen. Während bei monozentrischen Organismen die Zentromere und die Region direkt um die Zentromere wenige Gene vorweisen, sind diese bei holozentrischen Arten gleichmäßig über die gesamte Länge der Chromosomen verteilt. Bekannt ist, dass sich bei monozentrischen Arten die Chromosomen während der Interphase in hohem Maße miteinander vermischen, eine Eigenschaft, die bei der Regulierung der Genexpression eine Rolle zu spielen scheint. Bemerkenswerterweise waren diese weitreichenden Interaktionen in den Schnabelrieden mit Holozentromeren stark vermindert. Die Holozentrizität wirkt sich also grundlegend auf die Genomorganisation und das Verhalten der Chromosomen während der Zellteilung aus.
Bei holozentrischen Organismen beherbergt fast jedes Chromosomenfragment ein Zentromer und hat somit eine eigene Zentromerfunktion, was bei monozentrischen Arten nicht der Fall ist. Man geht davon aus, dass Holozentromere auf diese Weise Chromosomenfragmente und -fusionen stabilisieren und somit eine rasche Genomevolution ermöglichen, d. h. die Fähigkeit eines Organismus, rasche und umfassende Änderungen in der Anordnung der DNA vorzunehmen. Bei einem der von ihnen untersuchten Schnabelrieden konnten Marques und sein Team zeigen, dass die durch Holozentromere begünstigten Chromosomenfusionen es dieser Art ermöglichten, selbst nach der Vervierfachung des gesamten Genoms die gleiche Chromosomenzahl zu behalten. Bei einer anderen von ihnen untersuchten Schnabelriede, einer Art mit nur zwei Chromosomenpaaren, der niedrigsten Chromosomenanzahl aller Pflanzen, wurde festgestellt, dass die Holozentrizität für die drastische Verringerung der Chromosomenzahl verantwortlich ist. Holozentrische Chromosomen könnten also die Bildung neuer Arten durch schnelle Evolution auf Chromosomenebene ermöglichen.
Unsere Studie zeigt, so Marques, "dass der Übergang zu einer holozentrischen Chromosomenarchitektur die Art und Weise, wie Genome organisiert und reguliert werden, stark beeinflusst hat und dass sich Genome durch die Verschmelzung ihrer Chromosomen schnell weiterentwickeln können".
Die Ergebnisse des Teams zeigen vielversprechende Ansätze für die Pflanzenzüchtung, die in der Regel auf die Fähigkeit angewiesen ist, Gene zwischen Chromosomen und Organismen auszutauschen. "Holozentrische Pflanzen ermöglichen den Austausch von Genen in der Nähe der Zentromere, was bei monozentrischen Arten normalerweise unterdrückt wird. Wenn wir verstehen, wie holozentrische Pflanzen dies tun, könnten wir diese Gene in monozentrischen Arten „freischalten“ und sie für die Züchtung von leistungsfähigeren, widerstandsfähigeren Pflanzenarten zugänglich machen."
(Max-Planck-Institut für Pflanzenzüchtungsforschung)
Originalpublikation:
Hofstatter, Paulo G. et al.: Repeat-based holocentromeres influence genome architecture and karyotype evolution, Cell 2022, https://doi.org/10.1016/j.cell.2022.06.045