Auch genetisch identische Bakterien können sich in Form und Größe erheblich unterscheiden. Aber hat das funktionelle Auswirkungen? Forschende des Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI) und des Lehrstuhls für Mikrobiologie der Julius-Maximilians-Universität Würzburg (JMU) haben die funktionellen Unterschiede zwischen den verschiedenen Erscheinungsformen der Darmmikrobe Bacteroides thetaiotaomicron nun genauer untersucht. Dabei konnten sie zeigen, dass ein Zusammenhang zwischen Zelltyp und Stoffwechselfunktion besteht. Die Ergebnisse geben tiefere Einblicke in die mikrobielle Vielfalt des Darms und eröffnen neue Möglichkeiten für mikrobiombasierte Therapien. Die Studie wurde kürzlich im Fachjournal Cell Reports veröffentlicht.
Der menschliche Darm beherbergt eine Vielzahl an Bakterien. Diese spielen eine wichtige Rolle für unser Wohlbefinden, weil sie uns bei der Verdauung helfen und uns vor Krankheiten schützen. Dabei weisen die Mikroben eine sehr große Vielfalt auf, die über die bloßen Unterschiede zwischen einzelnen Arten hinausgeht. Sogar Bakterien derselben Art, mit gleicher genetischer Zusammensetzung, können sich in ihrem Aussehen unterscheiden.
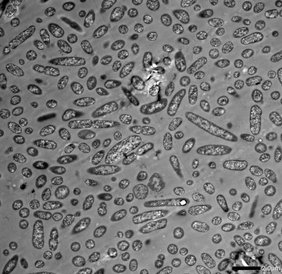
Ein gutes Beispiel ist die Gattung Bacteroides: Diese Bakterien gehören zu den häufigsten Darmbewohnern und verfügen über große morphologische Varianzen. Das bedeutet, sie kommen in verschiedenen Formen und Größen vor, auch Morphotypen genannt. Bisher war kaum erforscht, welche molekularen Mechanismen hinter dieser Formenvielfalt stecken und ob sie Auswirkungen auf die Funktion der Bakterien hat. Ein Team des Würzburger Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI), einem Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität Würzburg (JMU), sowie des Lehrstuhls für Mikrobiologie an der JMU hat die funktionellen Unterschiede zwischen den Erscheinungsformen von Bacteroides thetaiotaomicron genauer untersucht.
Form folgt Funktion
Die Analyse ergab, dass selbst innerhalb einer scheinbar einheitlichen Bakterienkultur deutliche Unterschiede in der Genaktivität bestehen – je nachdem, welcher Morphotyp vorliegt. „Viele dieser Gene sind dafür verantwortlich, dass Proteine produziert werden, die am Stoffwechsel beteiligt sind. Das deutet darauf hin, dass die morphologische Vielfalt auf eine Stoffwechselanpassung zurückzuführen ist“, erklärt Alexander Westermann, affiliierter Wissenschaftler am HIRI, Professor an der JMU und federführender Autor der Studie.
„Unsere Ergebnisse waren unerwartet“, ergänzt Élise Bornet, Doktorandin im Labor von Westermann und Erstautorin der Studie. „Sie zeigen, dass Unterschiede in der Zellgröße nicht bloß mit dem Zellzyklus zusammenhängen, also den Schritten der Zellteilung. Vielmehr scheint es sich um eine gezielte Strategie von Bacteroides thetaiotaomicron zu handeln, um sich besser an die dynamischen Bedingungen im Darm anzupassen.“
Eine neue Methode
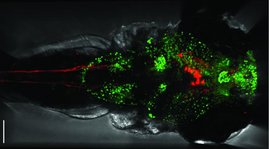
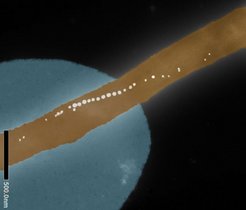
Zunächst haben die Forschenden die Bakterien entsprechend ihrer Größe getrennt und sortiert. Um deren Unterschiede in der Genaktivität zu charakterisieren, hat das Team in enger Zusammenarbeit mit der Arbeitsgruppe von Emmanuel Saliba am HIRI eine empfindliche RNA-Sequenzierungsmethode entwickelt. Diese Technologie macht es möglich, den Gehalt an Ribonukleinsäuren (RNAs, von engl. ribonucleic acids) in biologischen Proben zu bestimmen und zu analysieren. Darüber hinaus nutzten die Forschenden die Färbemethode Fluoreszenz-in-situ-Hybridisierung, mit der sie Boten-RNAs (mRNAs, von engl. messenger RNAs) direkt in der Zelle sichtbar machen können. Auf diese Weise konnte das Team Gene identifizieren, die als Marker für bestimmte Zellformen fungieren. Dabei fand es heraus, dass einige dieser Marker kausal die Zellgröße beeinflussen.
Ein Vorteil der neuen Methode ist, dass sie mit nur wenig Ausgangsmaterial auskommt. Im Grunde reichen bereits wenige Zellen aus. „Unser Protokoll ist nicht nur für unsere aktuelle Studie von Bedeutung, sondern stellt auch ein wertvolles Werkzeug für die Forschungsgemeinschaft dar“, sagt Westermann. „Unsere Technik kann zukünftig Forschenden dabei helfen, Unterschiede in der Genaktivität einzelner Zellen zu identifizieren – sowohl in Bacteroides als auch in anderen Mikrobiota-Arten“, fügt Bornet hinzu.
Der neue Analyseansatz könnte außerdem neue Möglichkeiten für die Medizin eröffnen: Da Bacteroides thetaiotaomicron wichtige Funktionen erfüllen, bildet ein tieferes Verständnis ihrer Vielfalt die Grundlage für neuartige mikrobiombasierte Therapien.
Kooperationen:
Diese Studie ist das Ergebnis einer Zusammenarbeit zwischen den Forschungsgruppen von Alexander Westermann, Lars Barquist, Emmanuel Saliba und Jörg Vogel am Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI), der Julius-Maximilians-Universität Würzburg (JMU), dem Helmholtz-Zentrum für Infektionsforschung (HZI) in Braunschweig, dem Leibniz Lungenzentrum in Borstel, der Stanford University in den Vereinigten Staaten von Amerika und der University of Toronto in Kanada.
Förderung:
Die Studie wurde durch den Europäischen Forschungsrat, das von der Deutschen Forschungsgemeinschaft (DFG) geförderte Center for Microbial Single-cell RNA-seq (MICROSEQ) der Universität Würzburg und die National Institutes of Health unterstützt. Des Weiteren wurde sie durch das DFG-Schwerpunktprogramm “Emergent Functions of Bacterial Multicellularity” (SPP 2389), das DFG-Graduiertenkolleg „3D-Infect“ (GRK2157) und den DFG-Sonderforschungsbereich DECIDE (SFB 1583) gefördert. Élise Bornet wurde durch das HIRI-Graduiertenprogramm „RNA & Infection” unterstützt. Kerwyn Casey Huang ist Chan Zuckerberg Biohub Investigator.
(Helmholtz-Zentrum für Infektionsforschung)
Originalpublikation:
Bornet E, Prezza G, Cecchino L, Jenniches L, Behrends J, Tawk C, Huang KC, Strowig T, Vogel J, Barquist L, Saliba AE, Westermann AJ:
Low-input RNA-seq suggests metabolic specialization underlying morphological heterogeneity in a gut commensal bacterium.
Cell Reports (2025), DOI: 10.1016/j.celrep.2025.115844