Jüngste Fortschritte im computergestützten Proteindesign beruhten hauptsächlich auf neuronalen Netzen und maschinellem Lernen zur Generierung von Bindern. Die Komplexität von Protein-Protein-Wechselwirkungen und die Einschränkungen datengesteuerter Modelle begrenzen jedoch den zukünftigen Fortschritt. Ein Forscherteam des Max-Planck-Instituts für Biologie Tübingen, der Universität Tübingen und des Universitätsklinikums Tübingen hat nun eine trainingsfreie computergestützte Pipeline entwickelt, die komplementäres Form-Matching nutzt, um die Erstellung von Proteinbindern zu steuern.
„Trotz signifikanter Fortschritte im computergestützten Proteindesign in den letzten Jahren bleibt das Design von Proteinbindern von Grund auf eine Herausforderung“, erklärt die Erstautorin der Studie, Kateryna Maksymenko. „Bisher setzen die erfolgreichsten Ansätze neuronale Netze ein. Unser Ziel war es, eine trainingsfreie Pipeline für das Binder-Design zu entwickeln. Wir wollten eine Design-Pipeline, die nicht nur die Erstellung ortsspezifischer Binder ermöglicht, sondern auch unser Verständnis der Proteinfaltung und -funktion vertieft.“
Vom Konzept zur praktischen Anwendung
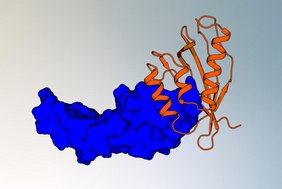
Diese Studie zeigt die erfolgreiche Anwendung des neuartigen Ansatzes zum Design von Proteinbindern, die auf zwei biologisch wichtige Moleküle abzielen: den Interleukin-7-Rezeptor alpha (IL-7Rα), der eine kritische Rolle bei Immunität und Leukämogenese spielt, und den Vascular Endothelial Growth Factor (VEGF), ein Schlüsselmolekül für die Angiogenese und ein therapeutisches Ziel bei verschiedenen Krankheiten.
Die Pipeline integriert die schnelle computergestützte Auswahl von formangepassten Gerüsten aus umfangreichen Proteindatenbanken mit einem physikbasierten Schnittstellendesign und Molekulardynamiksimulationen, um vielversprechende Binderkandidaten zu bewerten. Die besten Designs wurden dann experimentell validiert und zeigten eine starke Bindungsaffinität, hohe Stabilität und potente Aktivität in vitro und in vivo.
Eine Vision für die Zukunft des Protein-Engineerings
Diese neue Design-Pipeline vereinfacht nicht nur den Prozess, sondern bietet uns auch tiefere Einblicke in die physikalischen Grundlagen der Proteinfunktion. Darüber hinaus ermöglicht dieser Ansatz die Einbindung künstlicher Aminosäuren in entworfene Proteine. Die Forscher hoffen, dass diese Arbeit breitere Anwendungen inspirieren und die therapeutische Entdeckung beschleunigen wird.
Diese Arbeit ebnet den Weg für ein robustes, effizientes und interpretierbares Proteinbinder-Design, das möglicherweise die Arzneimittelentwicklung und molekulare Diagnostik transformieren wird.
Max-Planck-Institut für Biologie
Originalpublikation:
K. Maksymenko, V. Hatskovska, M. Coles, N. Aghaallaei, N. Pashkovskaia, N. Borbarán-Bravo, M. Pilz, P. Bucher, M. Volz, J. Pereira, M. D. Hartmann, G. Tabatabai, J. Feucht, S. Liebau, P. Müller, A. N. Lupas, J. Skokowa, M. ElGamacy, A Complementarity-Based Approach to De Novo Binder Design. Adv. Sci. 2025, e02015. https://doi.org/10.1002/advs.202502015