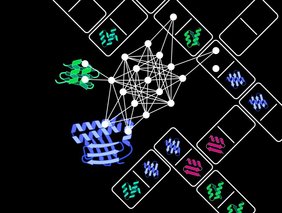
Proteine steuern auf Zellebene zahlreiche lebenswichtige Prozesse wie die Energiegewinnung oder die Vermehrung des Erbguts und werden daher häufig als „molekulare Maschinen“ der Zellen bezeichnet. Sie bestehen aus miteinander verknüpften Untereinheiten, den sogenannten Domänen. „Ähnlich wie Bauteile einer technischen Maschine haben diese Domänen spezifische Funktionen. Sie ermöglichen es den Proteinen beispielsweise, externe Signale wie Licht oder Temperatur zu erkennen oder chemische Reaktionen mithilfe von Proteinen zu katalysieren“, betont Prof. Niopek, der am IPMB die Abteilung Pharmazeutische Biologie leitet. Neuartige Proteine entstehen häufig auf natürliche Weise durch die Verbindung bereits existierender Domänen in neuen Kombinationen.
Inspiriert von diesen evolutionären Innovationsprozessen, haben Dominik Niopek und sein Team ein KI-Modell zur künstlichen Rekombination von Proteindomänen entwickelt. Für die Entwicklung und Optimierung dieser komplexen Computermodelle sind umfangreiche und qualitativ hochwertige Datensätze unverzichtbar. „Durch die automatisierte Analyse riesiger Proteindatenbanken konnten wir einen Datensatz mit über 100.000 Proteinen erstellen. Er bildet die enorme Vielfalt natürlich vorkommender Domänenkombinationen ab und war die zentrale Grundlage für das Training unseres KI-Modells“, so Benedict Wolf, Doktorand im Team von Prof. Niopek. Der Protein Domain Insertion Optimizer, kurz ProDomino, kann vorhersagen, welche Proteindomänen – ähnlich wie Dominosteine – passend zusammenfügt werden müssen, damit ein Fusionsprotein mit neuen Eigenschaften entsteht.
Mithilfe von ProDomino können zum Beispiel Proteinsensoren, die auf Reize wie Licht oder chemische Substanzen reagieren, mit sogenannten Effektorproteinen zur Steuerung zellulärer Prozesse kombiniert werden. So ist es den Heidelberger Wissenschaftlern gelungen, chemosensitive Domänen mit der Genschere CRISPR-Cas zu verbinden und dadurch künstliche CRISPR-Cas-Varianten zu erzeugen. Diese Hybridproteine lassen sich gezielt an- oder ausschalten und könnten somit die Sicherheit moderner Genomeditierungsverfahren deutlich erhöhen, wie Dr. Jan Mathony, Gruppenleiter in Prof. Niopeks Abteilung, erläutert. Die Forscherinnen und Forscher haben ihr KI-Modell bereits erfolgreich an verschiedenen Proteinen getestet und die Software als Open-Source-Tool der Wissenschaft zur Verfügung gestellt.
„Unser KI-Modell erlaubt es, künstliche Proteine nicht nur einfacher, sondern auch gezielter als bislang herzustellen. Das eröffnet neue Möglichkeiten, um die Präzision proteinbasierter Anwendungen in Biotechnologie und Medizin zu verbessern oder möglicherweise auch völlig neue Therapieansätze zu entwickeln“, so Prof. Niopek.
Universität Heidelberg
Originalpublikation:
Wolf, B., Shehu, P., Brenker, L. et al. Rational engineering of allosteric protein switches by in silico prediction of domain insertion sites. Nat Methods22, 1698–1706 (2025). doi.org/10.1038/s41592-025-02741-z