Mikrobiome prägen die Gesundheit von Menschen und Umwelt. Wie sie dies tun, das verrät eine Analyse der Funktionen, die einzelne Mikroben in diesen Gemeinschaften von Mikroorganismen übernehmen. Wissenschafter*innen der Universität Wien haben in Zusammenarbeit mit Photoniker*innen der Universität Boston eine Mikroskopie-Methode entwickelt, mit der sie Bakterienzellen in Millisekunden identifizieren und gleichzeitig ihre Stoffwechselaktivität bestimmen können. Ihre Methode zur Funktionsanalyse ist damit 100 bis 1.000 Mal schneller als bisher verfügbare Ansätze. Sie ermöglichte den Forscher*innen neue Erkenntnisse zum Abbau von Mukus, einem wesentlichen Bestandteil der Darmschleimhaut, durch das Darmmikrobiom des Menschen. Die Erkenntnisse können dazu beitragen, die Anfälligkeit einiger Menschen für entzündliche Darmerkrankungen zu erklären. Die Studie ist nun in der Fachzeitschrift „The Proceedings of the National Academy of Sciences (PNAS)“ erschienen.
Im menschlichen Darm gedeihen hunderte verschiedener Bakterienarten. Sie sind Teil des Darmmikrobioms und ihre vielfältigen Funktionen beeinflussen die Gesundheit des Menschen. Eine der wichtigsten Aufgaben der Mikrobiomforschung ist daher, die Funktionen unserer mikrobiellen Mitbewohner zu entschlüsseln und zu erforschen, wie diese gezielt beeinflusst werden können. Da sich Bakterien im Labor in Reinkultur häufig völlig unterschiedlich verhalten als in komplexen Lebensgemeinschaften, ist die direkte Funktionsanalyse, die die Funktionen einzelner Mikroben in komplexen Mikrobiomproben und damit in ihrem natürlichen Lebensraum erfasst, besonders aussagekräftig. In Zusammenarbeit mit der Arbeitsgruppe von Ji-Xin Cheng von der Universität Boston ist es Forscher*innen des Zentrums für Mikrobiologie und Umweltsystemwissenschaft der Universität Wien nun gelungen, einen bildgebenden Ansatz zu entwickeln, der Proben in Hochgeschwindigkeit analysiert.
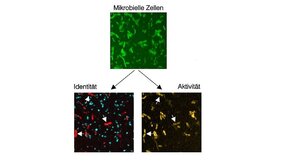
Die direkte Funktionsanalyse macht Bakterien und das Fressverhalten einzelner Bakterienzellen mittels chemischer Bildgebung sichtbar. Hierfür wird die Mikrobiomprobe mit einem Substrat versetzt, das mit einem stabilen (nicht radioaktiven) Isotop markiert ist. Mikroben, die dieses Substrat verstoffwechseln, werden entlang des Prinzips „Du bist was du isst“ mit dem stabilen Isotop markiert. Über den Isotopengehalt einzelner Bakterienzellen kann somit ihr Fressverhalten analysiert werden. Gleichzeitig werden unterschiedliche Bakterienarten farbig mit Hilfe fluoreszenzmarkierter Gensonden markiert, die sich jeweils an die Ribosomen ganz bestimmter Bakterien heften.
Aufgrund des hohen Zeitaufwands für die Messung selbst konnten Forscher*innen bisher sogar mit den modernsten Analyseverfahren immer nur relativ wenige Zellen pro Probe untersuchen. „Dies ist problematisch, da Mikrobiomproben hunderte Bakterienarten in verschiedenen Häufigkeiten enthalten. Die bisher verfügbaren Analysen waren daher nur für die jeweils dominanten Mikrobiommitglieder geeignet. Viele weitere gerieten aus dem Blick“, erklärt Michael Wagner, Mikrobiologe und stellvertretender Leiter des Zentrums für Mikrobiologie und Umweltsystemwissenschaft. Er initiierte die Zusammenarbeit mit den Bildgebungsexpert*innen aus den USA. Das internationale Forschungsteam reduzierte durch die Kombination von Stimulierter Raman Streumikroskopie (SRS) und Zwei-Photonen-Mikroskopie die für die Identifizierung und Bestimmung des Isotopengehalts notwendige Messzeit auf Millisekunden und damit um 2 bis 3 Größenordnungen im Vergleich zur konventionellen Ramanspektroskopie. „Mit dieser Hochgeschwindigkeitsmethode konnten wir nun in kürzester Zeit den Isotopengehalt von mehr als 30.000 Bakterienzellen aus menschlichen Darmproben nach Inkubation mit verschiedenen Zuckern, die im Darm-Mukus vorkommen, bestimmen“ berichtet Wagner.
„Dabei zeigte sich zu unserer Überraschung, dass bestimmte Clostridien – das ist eine Darmbakterienart – eine wichtige und bislang unbekannte Rolle beim Abbau des Schleimhautzuckers Fucose spielen“, ergänzt Fátima Pereira, Co-Erstautorin der Studie und Senior PostDoc in der Wiener Forschungsgruppe. Der Befund ist bemerkenswert, da Fucose die Schnittstelle zwischen der Schleimhaut und dem Darmmikrobiom darstellt. Jeder fünfte Mensch kann jedoch keine Fucosylierung des Darm-Mukus durchführen und zeigt ein erhöhtes Risiko für entzündliche Darmerkrankungen. Aktuelle Forschungsprojekte versuchen u.a. gezielt Probiotika einzusetzen, um das Erkrankungsrisiko dieser Personen zu reduzieren. Damit dies gelingen kann, ist es wichtig zu verstehen welche Mikroben vom Fucoseabbau im Darm profitieren und den betroffenen Personen möglicherweise fehlen.
In einem aktuellen Forschungsprojekt verwendet das Forschungsteam aus Wien und Boston die neu entwickelte Methode nun, um zu untersuchen, wie Medikamente, die zur Behandlung von Parkinson und Schizophrenie häufig verschrieben werden, das Darmmikrobiom des Menschen beeinflussen. „Der Einsatz dieser neuen Methode beschränkt sich aber nicht auf humane Mikrobiome, sondern eröffnet auch völlig neue Möglichkeiten in der Erforschung von Mikrobiomen der Umwelt“, erläutert Michael Wagner. Aufgrund dieses vielversprechenden Potentials habe das Zentrum für Mikrobiologie und Umweltsystemwissenschaft ein SRS-Fluoreszenzmikroskopie-Mikroskop angeschafft. Dieses in seiner für die Mikrobiomforschung optimierten Konfiguration einzigartige Großgerät in Europa wird gerade am Zentrum aufgebaut und ab Herbst 2022 für Forschungsprojekte interessierter Forscher*innen zur Verfügung stehen.
Universität Wien
Originalpublikation:
Gea X, Pereira FC, Mitteregger M, Berry D, Zhanga M, Hausmann B, Zhange J, Schintlmeister A, Wagner M, Cheng J-X: SRS-FISH: A high-throughput platform linking microbiome metabolism to identity at the single-cell level, 2022 - Proc Natl Acad Sci U S A, 119: e2203519119, DOI: https://doi.org/10.1073/pnas.2203519119