Regenwürmer leben im Boden in ständiger Wechselwirkung mit Bakterien, Pilzen und anderen Kleinstlebewesen. In ihrem Gewebe beherbergen sie symbiotische Mikroben oder kleine tierische Parasiten. Während die symbiotischen Bakterien größtenteils willkommene Gäste im Gewebe sind, kämpft das Immunsystem des Regenwurms gegen die Parasiten an. Diese wiederum versuchen den Angriffen standzuhalten. Durch die Kombination neuartiger Bildgebungsverfahren gelang es nun einem Forscherteam um Benedikt Geier vom Max-Planck-Institut für Marine Mikrobiologie (MPIMM) diese spannende Vielfalt an Interaktionen, die sich im Inneren des Regenwurms abspielen, abzubilden und in einem völlig neuen Licht zu betrachten.
Unser Verständnis der chemischen Interaktionen zwischen kleinen Tieren und Mikroorganismen, die sich in ihrem Körper aufhalten, ist extrem begrenzt. Diese Wissenslücke hat ihren Ursprung in einer methodischen Herausforderung: Um die grundlegenden Abläufe von Tier-Mikroben-Symbiosen aufzudecken, müssen wir verstehen welche chemischen Stoffe von den einzelnen Partnern wo produziert werden. Der Kern dieser Herausforderung ist es aber nicht nur bildlich darzustellen wie Moleküle auf Mikrometerebene verteilt sind. Es ist auch fast unmöglich, die chemischen Bilder bisheriger Methoden richtig zu interpretieren, wenn unbekannt ist, ob, wo und mit welchen nützlichen oder pathogenen Mikroben oder sogar tierischen Parasiten ein Gewebe infiziert ist.
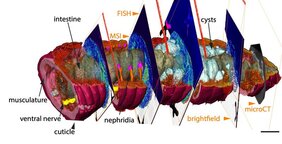
Eine mögliche Lösung bietet die neue Kombination hochauflösender Bildgebungsverfahren, wie das Forschungsteam nun gezeigt hat. „Wir stellen in unserer Studie die Chemo-Histotomografie vor, eine besondere dreidimensionale Darstellung von Chemie und Anatomie millimetergroßer Tiere und ihrer Parasiten auf zellulärer Ebene“, sagt Benedikt Geier, Erstautor der Untersuchung. „Diese Methode bietet eine neue Möglichkeit, Produkte des Stoffwechsels in Kleintiersymbiosen zu visualisieren und somit die Chemie dem tierischen Wirt und seinen mikrobiellen Partnern im Mikrometerbereich räumlich zuzuordnen.“
Die Chemo-Histotomografie, von den Forschenden kurz CHEMHIST genannt, besteht aus der Kombination von Mikro-Computertomografie und bildgebender MALDI-Massenspektrometrie. Die hochauflösende Mikro-Computertomografie ist nicht-invasiv und ermöglicht eine 3D Darstellung von Histologie, die aus einer Vielzahl von Röntgenbildern einer Probe rekonstruiert wird. Die bildgebende MALDI-Massenspektrometrie wiederum ist das derzeit leistungsfähigste Werkzeug, um mikrometergroße, natürliche Verteilungen von Stoffwechselprodukten zu visualisieren und um die chemischen Profile ihrem Produktionsort und möglicher Weise ihrem Produzenten zuzuordnen.
„Dieser Fortschritt ermöglichte es uns, einen Regenwurm aus der Umwelt zu nehmen und einen 3D-Atlas seiner chemischen und physikalischen Wechselwirkungen mit Mikroorganismen zu erstellen, die natürlich in seinem Gewebe vorkommen“, sagt Manuel Liebeke, Leiter der Forschungsgruppe Metabolische Interaktion und Betreuer der Studie. „Wichtig zu erwähnen ist hierbei auch, dass uns nicht nur die Biologie des Regenwurms interessiert. Eines unserer Hauptziele war es, CHEMHIST auch auf Tiere anwendbar zu machen, die direkt ihrem natürlichen Lebensraum entnommen wurden und essentiell für die Symbioseforschung sind.“ CHEMHIST übertrifft bisher vergleichbare Anwendungen, die für die medizinische Forschung an Mäusen entwickelt wurden, bei einer bis zu zwei Größenordnungen höheren Auflösung. Dies kann auch neue Türen für die Forschung an Insekten oder Korallen eröffnen, die an Land wie im Wasser Schlüsselmodelle der Symbioseforschung darstellen
Das Ganze ist mehr als die Summe der Teile
Mit der Kombination verschiedener in-situ-Bildgebungen in CHEMHIST deckten die Forschenden des MPIMM in Bremen Metabolite – also Produkte des Stoffwechsels – im Regenwurm auf, die Aufschluss geben könnten, wie er sich chemisch gegen Parasiten wehrt und wie diese sich wiederum gegen die Immunantwort des Regenwurmes schützen. Um jedoch überhaupt zu erkennen, dass es sich in den chemischen Bilddataten um Metabolite von Parasiten im Gewebe des Regenwurms handelt, war das anatomische 3D-Modell unabkömmlich. Ein hochauflösender Mikro-Computertomograph am Deutschen Elektronen Synchrotron in Hamburg ermöglichte es erst, die Parasiten im Gewebe zu identifizieren.
In Symbiosen ist der Austausch von Stoffwechselprodukten zwischen Tieren und ihren Mikroben bemerkenswerter Weise nicht auf symbiotische Gewebe beschränkt. Ein Beispiel ist die Darm-Hirn-Achse, bei der chemische Verbindungen im Darm von Mikroben produziert werden, die dann das Gehirn des Wirtes erreichen und somit grundlegende Prozesse beeinflussen könnten. Hier kann die nun vorgestellte Erweiterung der korrelativen chemischen 3D Bildgebung entscheidend sein, um die Verteilung von Metaboliten in solchen Symbiosen über Organe hinweg zu erfassen und somit eine Verbindung herzustellen, wie sich chemische Signale von Mikroben entscheidende Prozesse in ihrem Wirt beeinflussen. Die Methode kann also vielfältig eingesetzt werden und das Forschungsteam mit Benedikt Geier wendet sie gerade mit Tiefseemuscheln an.
„Da ich mich seit Jahren mit der 3D-Visualisierung von wirbellosen Tieren befasse, war es für mich besonders interessant, die Moleküle zu sehen, die sich hinter den morphologischen Strukturen verstecken“ sagt Bernhard Ruthensteiner, Leiter der Sektion Invertebrate Varia an der Zoologischen Staatssammlung München. Ermöglicht wurden die Ergebnisse durch eine interdisziplinäre Zusammenarbeit, die Wissenschaftler aus den Gebieten der Mikrobiologie, Zoologie, Chemie und Physik an einen Tisch gebracht hat. Dabei wurde schnell klar, dass eine Visualisierung wie der korrelative 3D-Atlas des Wurms die Wissenschaftskommunikation der Daten fördert.
Max-Planck-Institut für Marine Mikrobiologie
Originalpublikation:
Benedikt Geier; Janina Oetjen; Bernhard Ruthensteiner; Maxim Polikarpov; Harald Gruber-Vodicka; Manuel Liebeke: Connecting structure and function from organisms to molecules in small animal symbioses through chemo-histo-tomography, PNAS, Juni 2021
https://doi.org/10.1073/pnas.2023773118