Enzyme sind wichtige Biokatalysatoren in allen lebenden Zellen. Sie sind im Normalfall große Proteine, die kleinere Moleküle – sogenannte Substrate – an sich binden und diese anschließend in andere Moleküle umwandeln, die „Produkte“. Ohne die Enzyme könnte die Reaktion der Substrate in die Produkte nicht oder nur mit sehr kleiner Rate stattfinden. Die meisten Organismen besitzen Tausende verschiedene Enzyme. In vielen biotechnologischen Prozessen, aber auch im Alltag – von der Reifung von Brotteigen bis hin zu Waschmitteln – finden Enzyme vielfache Anwendungen.
Die maximale Geschwindigkeit, mit der ein bestimmtes Enzym seine Substrate in Produkte umwandeln kann, wird durch die sogenannte Wechselzahl kcat (englisch „turnover number“) bestimmt. Sie ist ein wichtiger Parameter für die quantitative Erforschung von Enzymaktivitäten und spielt eine entscheidende Rolle für das Verständnis des zellulären Stoffwechsels.
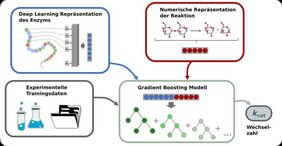
Allerdings ist es zeitaufwendig und teuer, die kcat-Werte experimentell zu bestimmen; daher sind ihre Werte für die allermeisten Reaktionen nicht bekannt. Die Arbeitsgruppe Computergestützte Zellbiologie an der HHU um Prof. Dr. Martin Lercher entwickelte nun ein neues Verfahren namens TurNuP, um kcat -Werte von Enzymen mithilfe von KI-Methoden vorherzusagen.
Um ein kcat -Vorhersagemodell zu trainieren, wurden mithilfe von Deep Learning-Modellen Informationen über die Enzyme und die katalysierten Reaktionen in numerische Vektoren umgewandelt. Diese numerischen Vektoren dienten als Eingabe eines Machine Learning-Modells – eines sogenannten Gradient Boosting-Modells –, das die kcat -Werte vorhersagt.
Erstautor Alexander Kroll: „TurNuP liefert bessere Ergebnisse als frühere Ansätze und kann sogar erfolgreich auf Enzyme angewendet werden, die nur eine geringe Ähnlichkeit zu den Enzymen im Trainingsdatensatz aufweisen.“ Mit bisherigen Modellen konnten keine sinnvollen Aussagen getroffen werden, wenn die Enzymsequenz nicht mindestens 40 Prozent mit denjenigen der Trainingsenzyme überstimmte. TurNuP dagegen macht schon bei solchen Enzymen sinnvolle Vorhersagen, bei denen die Übereinstimmung zwischen 0 und 40 Prozent liegt.
Prof. Lercher ergänzt: „Wir zeigen in unserer Studie, dass die Vorhersagen durch TurNuP verwendet werden können, um die Konzentrationen von Enzymen in lebenden Zellen deutlich genauer als bisher vorherzusagen.“
Um das Vorhersagemodell möglichst vielen Anwendern leicht zugänglich zu machen, entwickelte das HHU-Team einen benutzerfreundlichen Webserver, mit dem andere Forschende kcat-Werte von Enzymen vorhersagen lassen können.
Link zum Webserver: https://turnup.cs.hhu.de/
HHU
Originalpublikation:
Kroll, A., Rousset, Y., Hu, XP. et al. Turnover number predictions for kinetically uncharacterized enzymes using machine and deep learning. Nat Commun14, 4139 (2023). doi.org/10.1038/s41467-023-39840-4