Mikroorganismen spielen eine entscheidende Rolle bei der Pflanzenernährung, beeinflussen die Gesundheit der Pflanzen, stärken ihre Toleranz gegenüber Stressfaktoren wie Trockenheit und unterstützen die Abwehr von Krankheitserregern. Die Nutzung dieser mikrobiellen Partnerschaften könnte einen bedeutenden Beitrag zu einer nachhaltigeren Landwirtschaft leisten, die weniger auf Düngemittel und Pestizide angewiesen ist.
Das Verständnis der Mechanismen, mit denen Mikroorganismen ihre Wirte besiedeln, ist Voraussetzung für die Entwicklung und Anwendung von Mikrobiomen mit nützlichen Funktionen. Daher untersuchen Forschende häufig, wie einzelne Mikroorganismen mit Pflanzen interagieren. Die genaue Art und Weise, wie mehrere Mikroben gleichzeitig Pflanzen besiedeln und mit ihnen interagieren, damit komplexere Wirt-Kommensal-Beziehungen entstehen können, blieb bisher weitgehend unbekannt. Dies ist frustrierend, da Informationen über die Interaktion einer einzelnen Mikrobe mit ihrer Wirtspflanze nicht zwangsläufig repräsentativ für die komplexe Realität einer mikrobiellen Gemeinschaft sind. Die Herausforderung war im Wesentlichen technischer Natur - wie kann man das Verhalten einzelner Stämme in einem Heuhaufen von Mikroben und der Pflanze selbst präzise charakterisieren?
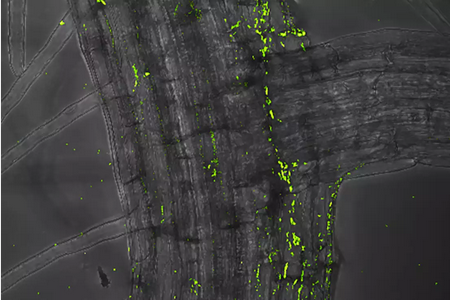
Genaktivität während der Besiedelung
Um das Problem anzugehen, untersuchten Nathan Vannier, Erstautor der Studie, und Stéphane Hacquard die Besiedlung einzelner Mikroben auf den Wurzeln von Pflanzen in komplexen Gemeinschaften. Ihr Ansatz begann mit mikrobenfreien Ackerschmalwandpflanzen. Anschließend führten sie eine definierte Gemeinschaft von Mikroben ein, die die beobachtete Vielfalt an Pflanzenwurzeln in der Natur repräsentierte. Ziel war es zu untersuchen, wie diese Mikroben ihren Wirt besiedeln. Da sie die Identität dieser Bakterien kannten und über Referenzsequenzen für ihr genetisches Material verfügten, konnten sie charakterisieren, welche Gene während der Pflanzenbesiedlung aktiviert oder unterdrückt wurden.
Diese Analyse ermöglichte es ihnen, zahlreiche Gene zu identifizieren, die bei vielen verschiedenen Bakterien in Wurzeln stark exprimiert wurden, und die möglicherweise an der Besiedlung des Wirts beteiligt sind. Ein Gen reguliert bakterielle Virulenz und Stressreaktionen, ein anderes ist am Transport von Transmembranpolymeren beteiligt und eine Gruppe von Genen agiert gemeinsam als Phosphatsensor. Die Mutation von drei dieser Gene in Bakterien beeinträchtigte deren Fähigkeit, Wurzeln zu besiedeln, ohne ihr Wachstum zu beeinflussen. Der Ansatz der Autoren ermöglichte es ihnen, eine zentrale Gruppe von Genen zu identifizieren, die viele Bakterien benötigen, um auf Pflanzenwurzeln zu überleben.
Die von Hacquard und seinem Team angewandte Strategie ermöglichte es, sowohl die strukturelle als auch die funktionelle Organisation komplexer mikrobieller Gemeinschaften zu verstehen, die Pflanzenwurzeln besiedeln. Die identifizierten Gene werden möglicherweise von sehr unterschiedlichen Bakterien genutzt, um sich auf ihren Wirten anzusiedeln und dort zu überleben. "Unsere Ergebnisse könnten möglicherweise den Weg für die Entwicklung nützlicher Bakterien ebnen, die Pflanzen besiedeln und deren Gesundheit fördern können. Dies hat nicht nur Auswirkungen auf eine nachhaltige Landwirtschaft, sondern auch auf Fortschritte in der medizinischen Forschung", betont Stéphane Hacquard.
Max-Planck-Institut für Pflanzenzüchtungsforschung
Originalpublikation:
Vannier, N., Mesny, F., Getzke, F. et al. Genome-resolved metatranscriptomics reveals conserved root colonization determinants in a synthetic microbiota. Nat Commun14, 8274 (2023). doi.org/10.1038/s41467-023-43688-z