Ein hochentwickelter SNAP-tag-Mutant
Selbstmarkierende Protein-Tags wie SNAP-tag bieten die Möglichkeit, helle und fotostabile synthetische Fluorophore an Proteine von Interesse anzubinden, um diese später zu analysieren. Sie sind in der Biochemie weit verbreitet.
„SNAP-tag wurde ursprünglich aus einem menschlichen DNA-Reparaturenzym entwickelt. Ein bereits stark verändertes Protein weiter zu verändern, war eine der größten Herausforderungen in unserer Arbeit“, sagt Stefanie Kühn, die das Projektteam leitete. Diesem gehörten weitere Wissenschaftler*innen des Max-Planck-Instituts (MPI) für medizinische Forschung an, darunter Julien Hiblot, Veselin Nasufovic und Direktor Kai Johnsson, ebenso ein Team der Universität Groningen. „Außerdem wollten wir einen Substratkern finden, der in Zellen mit verschiedenen Fluorophoren gut funktioniert. Wir haben uns für eine Kombination aus Substratoptimierung und Protein-Engineering entschieden, um SNAP-tag2 zu entwickeln.“
Erhöhte Reaktionsfähigkeit
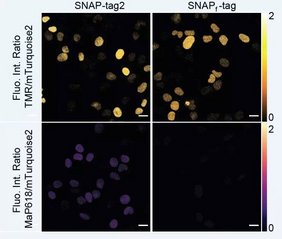
Die bisher weit verbreitete Verwendung von SNAP-tag bei der Arbeit mit lebenden Zellen kann dadurch eingeschränkt sein, dass sie Prozesse vergleichsweise langsam markieren und ihre Substrate nur begrenzt zelldurchlässig sind. Das Ziel der Wissenschaftler*innen am MPI für medizinische Forschung war es, eine Lösung für dieses Problem zu finden. SNAP-tag2 reagiert schnell mit den neuen zelldurchlässigen Substraten: Es zeigt eine Markierungsrate, die gegenüber vergleichbaren SNAP-tag-Substrat-Paaren einhundertfach verbessert ist.
Effizientere Fluoreszenzmarkierung in lebenden Zellen
SNAP-tag2 bietet weitere Vorteile. Wird es mit stark fluoreszierenden Farbstoffen markiert, zeigt es im Vergleich zum derzeit verwendeten SNAP-tag eine um das Fünffache hellere Fluoreszenz. SNAP-tag2 führt auch zu besseren Ergebnissen als bisherige Tools, wenn es in Säugetierzellen und in Hefe eingesetzt wird. Außerdem ermöglicht es den Einsatz von superauflösenden Mikroskopietechniken wie der STED-Mikroskopie sowie die Arbeit mit anderen Zelltypen, die als unempfindlich gegenüber chemischer Markierung bekannt sind, wie beispielsweise Hefezellen.
„SNAP-tag2 ist zusammen mit seinen verbesserten Substraten in allen von uns getesteten Anwendungen den bisher verwendeten SNAP-tag-Versionen überlegen. Wir hoffen, dass es sich auch in In-vivo-Anwendungen bewähren wird“, sagt Stefanie Kühn.
Weitere Anwendungsbereiche möglich
Zusammenfassend geht das Team davon aus, dass die verbesserte Performance von SNAP-tag2 dazu beiträgt, den Einsatz dieses bereits weit verbreiteten Tools für die Echtzeit-Zell-Bildgebung und andere Anwendungen in den Lebenswissenschaften weiter auszubauen. Darüber hinaus eröffnet SNAP-tag2 neue Möglichkeiten für Mehrfarben-Bildgebungsanwendungen.
Max-Planck-Institut für medizinische Forschung
Originalpublikation:
Kühn, S., Nasufovic, V., Wilhelm, J. et al. SNAP-tag2 for faster and brighter protein labeling. Nat Chem Biol (2025). doi.org/10.1038/s41589-025-01942-z