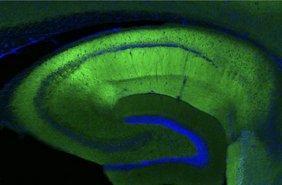
Forschende am Max-Planck-Institut für Hirnforschung haben die molekulare Landschaft des Hippokampus von Mäusen kartiert, einer Region, die für das Lernen und Gedächtnis von zentraler Bedeutung ist. Mithilfe einer Kombination von RNA- und Proteinprofilierung mit neuen Methoden zur Isolierung von Hirnregionen und Synapsen identifizierte das Team Tausende von Molekülen mit unterschiedlichen räumlichen Mustern. Die in Nature Communications veröffentlichte Studie zeigt, wie die lokale Proteinproduktion und die synaptische Vielfalt die Funktion des Hippokampus beeinflussen, und stellt der Forschungsgemeinschaft eine frei zugängliche Ressource zur Verfügung.
Um die Komplexität des Gehirns zu verstehen, sind detaillierte, quantitative Informationen sowohl für Boten-RNAs (mRNAs) als auch für Proteine über Regionen, Zelltypen und Kompartimente hinweg erforderlich. Während in bisherigen Studien entweder Transkripte oder Proteine in bestimmten Hirnarealen vermessen wurden, haben nur wenige Studien beide Molekülklassen systematisch parallel integriert. In ihrer neuen Arbeit präsentieren Dr. Eva Kaulich, Quinn Waselenchuk und Kolleg*innen aus dem Schuman-Labor am Max-Planck-Institut für Hirnforschung in Frankfurt deshalb den ersten integrierten Transkriptom- und Proteom-Atlas des Hippokampus von Mäusen mit synaptischer Auflösung. Der Hippokampus, eine für das Lernen und Gedächtnis zentrale Region, wurde aufgrund seiner gut charakterisierten Struktur, Funktion und Konnektivität ausgewählt.
Die Forschenden kombinierten die Methoden der RNA-Sequenzierung (RNA-seq) und der Flüssigchromatographie-Tandem-Massenspektrometrie (LC-MS/MS) mit präziser Mikrodissektion von Hippokampus-Subregionen und -Schichten sowie mit Fluoreszenz-aktivierter Synaptosomen-Sortierung (FASS). Mithilfe dieser Methoden gelang es, mehr als 17.000 mRNAs und 10.000 Proteine systematisch zu vermessen und unterschiedlichen Anreicherungsmustern aufzudecken, z.B. für Rezeptoren, Ionenkanäle, Adhäsionsmoleküle und Stoffwechselregulatoren.
Die Integration der Datensätze offenbarte komplexe regulatorische Beziehungen. Während einige Proteine ihre mRNA-Levels genau widerspiegelten, war dies bei anderen nicht der Fall. Dies deutet auf eine zusätzliche Kontrolle durch die Halbwertszeit der Proteine und die lokale Translation hin. Die Analyse der neuronalen Kompartimente zeigte außerdem, dass distale Dendriten wahrscheinlich stärker auf lokale Proteinsynthese angewiesen sind, um ihre molekulare Identität aufrechtzuerhalten.
„Unser Atlas bietet einen beispiellosen Einblick in die Organisation von RNA und Proteinen im Hippokampus. Dies eröffnet neue Möglichkeiten, um zu untersuchen, wie molekulare Vielfalt die Gehirnfunktion und Plastizität unterstützt“, sagt Co-Erstautorin Eva Kaulich, Postdoktorandin im Schuman-Labor. „Durch die Integration von Transkriptom- und Proteomdaten mit subzellulärer Auflösung können wir nun besser verstehen, wie Neuronen ihre molekulare Maschinerie lokal regulieren, insbesondere an den Synapsen“, fügt Co-Erstautor Quinn Waselenchuk hinzu. Er ist Doktorand in den Laboren von Erin Schuman und Julian Langer an den Max-Planck-Instituten für Hirnforschung und Biophysik.
„Der Hippokampus dient seit langem als Modellsystem in den Neurowissenschaften. Mit der Veröffentlichung dieses Atlas möchten wir der Fachwelt ein Werkzeug an die Hand geben, mit dem sie untersuchen kann, wie die molekulare Organisation die neuronale und synaptische Funktion beeinflusst“, fasst Erin Schuman zusammen.
Der Atlas ist öffentlich zugänglich unter syndive.org.
Max-Planck-Institut für Hirnforschung
Originalpublikation:
Kaulich, E., Waselenchuk, Q., Fürst, N. et al. An integrated transcriptomic and proteomic map of the mouse hippocampus at synaptic resolution. Nat Commun16, 7942 (2025). doi.org/10.1038/s41467-025-63119-5