Mit der hochauflösenden Mikroskopie ist es theoretisch möglich, Zellstrukturen mit einer Auflösung von wenigen Nanometern abzubilden. Doch in der Praxis gelang das bisher nicht.
Der Grund dafür: Zur Markierung der Zellstrukturen werden meistens Antikörper eingesetzt, die einen fluoreszierenden Farbstoff tragen. Der Farbstoff befindet sich darum nicht direkt an der Zielstruktur, sondern rund 17,5 Nanometer davon entfernt. Unter anderem wegen dieses Abstandsfehlers konnte die theoretisch erreichbare Auflösung bislang nicht realisiert werden.
Ein internationales Forschungsteam hat diese Hürde jetzt überwunden. Das gelang durch die Kombination der hochauflösenden Fluoreszenzmikroskopie dSTORM mit der Expansionsmikroskopie ExM. Das Fachjournal Nature Communications stellt die Ergebnisse vor.
Federführend bei der Publikation war ein Team vom Biozentrum der Julius-Maximilians-Universität (JMU) Würzburg: Professor Markus Sauer, Leiter des Lehrstuhls für Biotechnologie und Biophysik, mit den Doktoranden Fabian Zwettler und Sebastian Reinhard. Maßgeblich beteiligt waren auch die Professoren Paul Guichard von der Universität Genf (Schweiz) und Toby Bell von der Monash University (Australien).
Hemmnisse bei der Kombination von dSTORM und ExM
Mit der Methode dSTORM, die in der Arbeitsgruppe von Professor Sauer entwickelt wurde, gelingt eine fast molekulare Auflösung von rund 20 Nanometer. Um die Auflösung weiter zu steigern, schien eine Kombination mit der seit wenigen Jahren verfügbaren Expansionsmikroskopie aussichtsreich.
Bei der ExM wird die zu untersuchende Probe in ein quellbares Polymer vernetzt. Dann zerstört man die Wechselwirkungen der Moleküle in der Probe und lässt diese mit Wasser aufquellen. Dadurch kommt es zu einer Expansion: Die abzubildenden Moleküle driften um den Faktor vier räumlich auseinander.
Warum sich die beiden Methoden bisher nicht kombinieren ließen:
• Die Fluoreszenzfarbstoffe, die bei dSTORM zu Markierung der Moleküle verwendet werden, überlebten die Polymerisation des wässrigen Gels nicht.
• Für dSTORM ist eine Pufferlösung nötig, die aber die expandierte Probe sich wieder zusammenziehen ließ.
Abstandsfehler deutlich verringert
„Durch eine Stabilisierung des Gels und eine Immunfärbung erst nach der Expansion könnten wir diese Hürden überwinden und die beiden Mikroskopiemethoden erfolgreich miteinander verbinden“, freut sich Markus Sauer. Dadurch schmelze der Abstandsfehler bei 3,2-facher Expansion auf nur noch fünf Nanometer. Das mache erstmals ein Fluoreszenzimaging mit molekularer Auflösung möglich.
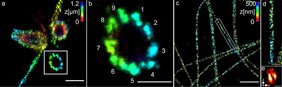
Wie gut ihre Methode funktioniert, zeigten die Forscher an Zentriolen und Strukturen, die aus dem Protein Tubulin aufgebaut sind. Unter anderem konnten sie Tubulinröhrchen als hohle Zylinder mit einem Durchmesser von 25 Nanometern visualisieren. An den Zentriolen gelang es, Dreiergruppen aus Tubulinstrukturen mit einem Abstand von 15 bis 20 Nanometer scharf abzubilden. Die Zentriolen sind Zellstrukturen, die bei der Zellteilung eine wichtige Rolle spielen.
Professor Sauers Fazit: „Bei vielen wichtigen Zellkomponenten können wir durch die Kombination von ExM und dSTORM nun erstmals detaillierte Einblicke in die molekulare Funktion und Architektur bekommen.“ Das Team will darum als nächstes die Methode bei verschiedenen Strukturen, Organellen und Multiproteinkomplexen der Zelle anwenden.
Universität Würzburg
Originalpublikation:
Zwettler, F.U., Reinhard, S., Gambarotto, D. et al. Molecular resolution imaging by post-labeling expansion single-molecule localization microscopy (Ex-SMLM). Nat Commun 11, 3388 (2020).