Vorwissen über den Stammbaum mit auf den Weg geben
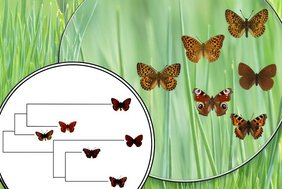
„Die meisten bisherigen KI-Algorithmen tun sich schwer damit, biologische Daten im Licht der Evolution zu analysieren, weil sie nicht wissen, worauf sie achten sollen und sich durch zufällige Muster in die Irre führen lassen”, so Axel Mosig. Das Vorwissen über die Abstammungsstammbäume der zu analysierenden Arten haben die Bochumer Forschenden ihrer KI mit auf den Weg gegeben. Der Ansatz beruht darauf, beim Trainieren der KI jeweils Vierergruppen von Spezies in den als korrekt angenommenen Abstammungsbaum einzuordnen. Darin liegen die Informationen über je nahe und entfernte Verwandtschaften vor. „Wenn alle Vierergruppen evolutionär richtig eingeordnet sind, lässt sich daraus der ganze Abstammungsbaum wie in einem Puzzle zusammensetzen”, erläutert Luis Hack, der ebenfalls an der Studie mitgewirkt hat. So kann die KI in den Sequenzen Muster aufdecken, die entlang dieses Abstammungsbaums evolviert sind”.
Der Clou: Die Methode funktioniert nicht nur für genetische Sequenzdaten, sondern auch für beliebige andere Arten von Daten, beispielsweise Bilddaten oder Strukturmuster von Biomolekülen aus unterschiedlichen Spezies. Nachdem die RUB-Bioinformatikerinnen und Bioinformatiker den Ansatz in der aktuellen Arbeit zunächst für DNA-Sequenzdaten etabliert haben, arbeiten sie bereits an Anwendungen auf Bilddaten. „Man könnte dann beispielsweise hypothetische Bilder von evolutionären Vorgängerspezies, rekonstruieren”, deutet Hack ein Potenzial der Methode für zukünftige Arbeiten an.
Ruhr-Universität Bochum
Originalpublikation:
Vivian B. Brandenburg, Ben Luis Hack, Axel Mosig: A quartet-based approach for inferring phylogenetically informative features from genomic and phenomic data, in: Computational and Structural Biotechnology Journal, 2025, DOI: 10.1016/j.csbj.2025.08.015, https://www.csbj.org/article/S2001-0370(25)00337-X/fulltext