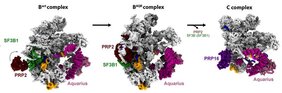
Damit eine Zelle Proteine produzieren kann, muss sie zunächst die in unseren Genen kodierten Bauanleitungen in eine lesbare Form bringen. Dafür wird das Gen in eine Rohfassung der Boten-RNA (mRNA) übersetzt. Diese prä-mRNA enthält die Bauanleitung allerdings noch nicht an einem Stück. In einem komplizierten Prozess muss das Spleißosom nicht benötigte Abschnitte herausschneiden und die informationstragenden Abschnitte, die Exons, neu zusammensetzen. Dieser Vorgang wird Spleißen genannt. Spleißen hat einen entscheidenden Vorteil: Bei Bedarf können die Exons unterschiedlich zusammengesetzt werden. Beim Menschen liefern so etwa 20.000 Gene die Bauanleitungen für mehr als 100.000 verschiedene Proteine.
„Das Spleißosom gilt als komplexeste und dynamischste molekulare Maschine in unseren Zellen. Es besteht unter anderem aus über 150 Proteinen“, erklärt Vladimir Pena, der die Forschungsarbeiten zunächst als Gruppenleiter am MPI für Multidisziplinäre Naturwissenschaften und dann als Professor am ICR leitete. „Beim Spleißen durchläuft das Spleißosom zahlreiche Schritte, in denen es seine Struktur und Zusammensetzung verändert. Diese Schritte werden durch molekulare Motoren, die Helikasen, angetrieben. Der komplizierte Aufbau des Spleißosoms macht es sehr schwierig, die Funktion der Maschine im Detail zu untersuchen und zu verstehen.“
Mitten ins Herz des Spleißosoms
Mithilfe der Kryo-Elektronenmikroskopie und biochemischer Methoden konnte das Team um Pena das Spleißosom in fast atomarer Auflösung mitten im Anschaltprozess „einfangen“. Im Fokus dieses Vorgangs steht eine Komponente des Spleißosoms, SF3B1 genannt, die für die Aktivierung der Maschine unerlässlich ist.
„Wir haben entdeckt, dass das Spleißosom in Menschen nur mithilfe von zwei Helikasen, PRP2 und Aquarius, gestartet werden kann, indem sie SF3B1 verändern“, so Pena. „Helikasen sind eine besondere Art von Proteinen. Sie wandeln die in einem ATP – ein chemisches Molekül, das in lebenden Zellen Energie bereitstellt – gespeicherte chemische Energie in mechanische Arbeit um. Sie sind also autonome Motoren, die von der ‚ATP-Batterie‘ angetrieben werden“, erläutert der Wissenschaftler.
Wie die Forschenden weiterhin zeigen konnten, wechselwirkt PRP2 mit SF3B1 auf eine Art und Weise, wie sie nie zuvor bei Helikasen beobachtet wurde. Constantin Cretu, einer der Erstautor*innen der Studie, beschreibt die Besonderheit: „Anstatt wie andere Proteine an die Außenseite des Spleißosoms zu binden, wandert PRP2 entlang des zu bearbeitenden RNA-Strangs bis zum ‚Herzen‘ des Spleißosoms. Dabei ordnet es dessen Struktur und Zusammensetzung um und versetzt die molekulare Maschine in einen aktiven Zustand.“ Die Wissenschaftler*innen gehen davon aus, dass auch andere Helikasen auf diese neue und unerwartete Weise arbeiten könnten. Im Gegensatz zu PRP2 verrichtet Aquarius seine Arbeit von der Oberfläche des Spleißosoms.
„Mit unseren Experimenten haben wir einen entscheidenden neuen Zwischenschritt im Zyklus des Spleißosoms sichtbar gemacht, der bisher noch nicht beschrieben wurde“, sagt Jana Schmitzová, ebenfalls Erstautorin der Publikation. „Dadurch haben wir wichtige Neuordnungen der Proteine aufgedeckt: Einige werden aussortiert und ersetzt, andere ändern ihre Position. Es ist faszinierend zu sehen, wie alle Proteinakteure auf solch orchestrierte Weise miteinander wechselwirken.“
Fehler beim Spleißen triggern Krebs
SF3B1 ist der Bestandteil des Spleißosoms, der bei einigen Krebserkrankungen wie Leukämie, Aderhautmelanom, Bauchspeicheldrüsen- und Prostatakrebs am häufigsten Mutationen aufweist. Diese Komponente des Spleißosoms ist daher ein wichtiges Ziel für Antitumormedikamente. „Wir hoffen, dass unsere Erkenntnisse neue Forschungsarbeiten anregen, die die Ursachen der Krebsentstehung weiter aufklären. Diese können dazu beitragen, Krebsmedikamente zu entwickeln, die direkt auf den Spleißvorgang abzielen“, so Pena.
Max-Planck-Institut für Multidisziplinäre Naturwissenschaften
Originalpublikation:
Schmitzová, J., Cretu, C., Dienemann, C., Urlaub, H., & Pena, V.: Structural basis of catalytic activation in human splicing. Nature 617, 1-9 (2023). https://doi.org/10.1038/s41586-023-06049-w