In der aktuellen Studie, die von Eduard Unterauer in Jungmanns Labor am MPI für Biochemie und der LMU geleitet wurde, stellen die Forscher SUM-PAINT vor. Dabei handelt es sich um eine technologische Neuentwicklung in der Superauflösungsmikroskopie, die nun erstmals eine sehr schnelle und praktisch unbegrenzte Visualisierung und Kartierung einer Vielzahl von Proteinen ermöglicht.
„Die Komplexität lebender Systeme reicht von ganzen Organismen und Geweben über den Aufbau komplexer zellulärer Netzwerke bis hin zur Organisation und Interaktion einzelner Biomoleküle. Damit wir diese Komplexität in ihrer Gesamtheit verstehen können müssen sowohl der Ort, als auch Identität und Interaktion einzelner Biomoleküle gleichzeitig untersucht werden. Man nennt solche Methoden, die mehrere Signale bündeln, Multiplexing-Methoden. Für ein umfassendes Verständnis der Proteinorganisation müssen insgesamt vier kritische Herausforderungen bewältigt werden: Empfindlichkeit, Durchsatz, räumliche Auflösung und Multiplexing-Fähigkeit,“ erklärt Eduard Unterauer, Ko-Erstautor der Studie.
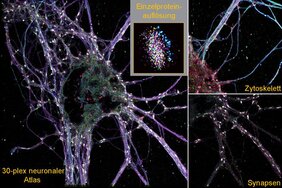
Das Team konzentrierte sich auf das komplexe Umfeld neuronaler Zellen des Gehirns und erstellte - zum ersten Mal überhaupt - einen neuronalen Atlas mit Einzelmolekülauflösung für 30 verschiedene Proteinarten. Dank des verbesserten Durchsatzes und der Multiplexing-Möglichkeiten konnten sie die Komplexität der synaptischen Proteinzusammensetzung von fast 900 einzelnen Synapsen entschlüsseln.
Um diese umfangreichen Datensätze weiter im Detail zu untersuchen, entwickelte das Forschungsteam eine durch maschinelles Lernen gestützte Analysepipeline. Durch die Auswertung von 1600 Merkmalen aus den Bildgebungsdatensätzen, wie beispielsweise Proteingehalt, Verteilung oder Form, entdeckten die Wissenschaftler*innen einen bisher unbekannten Typ chemischer Synapsen. Diese Synapsen machen nur etwa 1 % aller Synapsen aus und wären mit anderen bildgebenden Verfahren nicht entdeckt worden.
Mit SUM-PAINT stellt das Team einen integrierten Workflow für die Datenerzeugung und -analyse bereit, der von Forscher*innen auf der ganzen Welt eingesetzt werden kann. SUM-PAINT kann auf relativ einfache Weise mit handelsüblichen Mikroskopen eingesetzt werden.
„Wir sind davon überzeugt, dass SUM-PAINT nicht nur ein Meilenstein auf dem Weg zur Entschlüsselung der Komplexität der Zellbiologie auf molekularer Ebene darstellt, sondern einen potentiellen Durchbruch bei der Entdeckung neuer therapeutischer Ansätze neurodegenerativer Krankheiten ermöglichen kann,“ sagt Ralf Jungmann, Leiter der Forschungsgruppe Molekulare Bildgebung und Bionanotechnologie am MPI für Biochemie und Inhaber des Lehrstuhls für Molekulare Physik des Lebens an LMU.
Indem SUM-PAINT einen detaillierten Blick auf den Ort und die Interaktion einer großen Anzahl von Proteinen auf molekularer Ebene ermöglicht, eröffnet es ungeahnte Möglichkeiten bisher verborgene Details neurologischer Störungen zu untersuchen. Dadurch könnte die neue Methode zu einem tieferen Verständnis zugrunde liegender Mechanismen von Krankheiten, wie Parkinson oder der Alzheimer-Demenz, beitragen.
(Max-Planck-Institut für Biochemie)
Originalpublikation:
Eduard M. Unterauer*, Sayedali Shetab Boushehri*, Kristina Jevdokimenko*, Luciano A. Masullo, Mahipal Ganji, Shama Sograte-Idrissi, Rafal Kowalewski, Sebastian Strauss, Susanne C.M. Reinhardt, Ana Perovic, Carsten Marr, Felipe Opazo, Eugenio F. Fornasiero# and Ralf Jungmann#, Spatial proteomics in neurons at single-protein resolution. Cell, March 2024