Bei der sexuellen Fortpflanzung von Organismen werden die Gene der Eltern neu kombiniert, man spricht von „Rekombination“. Das Genom der Nachkommen ist quasi ein Mosaik aus den Erbanlagen der Eltern; aus deren Genen ergibt sich eine neue Gesamtheit ganz individueller Eigenschaften der Nachkommen: deren jeweiliger „Phänotyp“.
Um neue Pflanzensorten zu züchten, ist es wünschenswert, möglichst viele Kombinationsmöglichkeiten zu haben. Dies ist unter anderem dann gegeben, wenn nicht ganze DNA-Stränge vererbt werden, sondern kürzere Abschnitte. Die Eigenschaft in den Nachkommen solcher Pflanzen zu erzeugen heißt „hochrekombinogen“.
Eine solche Eigenschaft könnte mittels gentechnischer Eingriffe künstlich in das Pflanzengenom eingefügt werden. Ein Team von Biologinnen und Biologen vom HHU-Institut für Quantitative Genetik und Genomik der Pflanzen verfolgte aber einen anderen Weg: Sie entwickelten eine Methode, die entsprechende hochrekombinogen Individuen unter natürlich vorkommenden Varianten erkennt.
Dazu Federico A. Casale, Doktorand am Institut und Erstautor der Studie: „Die Idee, Pflanzen mit einer natürlich auftretenden hohen Rekombinogenität zu selektieren, gab es schon vor 70 Jahren. Nur praktisch gelang dies bisher nicht, weil es experimentell zu aufwändig war, von einer sehr großen Zahl von Individuen diese Eigenschaft zu bestimmen, um die wenigen hochrekombinogen Individuen zu finden.“
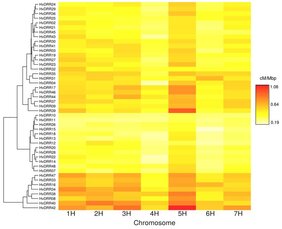
Die Düsseldorfer Forschenden arbeiteten mit 45 Familien der Sommergerste mit jeweils rund 100 Familienmitgliedern. Sie untersuchten in deren Nachkommenschaft rund 50.000 sogenannte molekulare Sonden und stellten so fest, wie sich die Genome der Eltern in den Nachkommen mischten. Vor allem sahen sie, dass sich der Mischungsgrad – die Variabilität der Mosaike zwischen verschiedenen Familien – um das Zweieinhalbfache unterscheiden kann.
Sie fanden weiterhin, dass eine hohe Variabilität in 90 Prozent der Fälle auf ein Elternteil zurückgeführt werden kann – ein klares Indiz, dass bei diesem Individuum die gesuchte hohe Rekombinogenität vorliegt und vererbt wurde. Mit dieser Kenntnis kann gezielt auf diese Eigenschaft hin gezüchtet werden.
Desweitern ermittelten die Forschenden durch eine Computersimulation, dass die Unterschiede in der Rekombinogenität mit rund 80 Prozent Genauigkeit vorhergesagt werden können. Die Düsseldorfer Methode erlaubt es, Selektionsprogramme für die Züchtung erheblich effizienter zu gestalten und somit schneller Pflanzen- und auch Tierarten zu entwickeln, die gewünschte Eigenschaften haben, ohne dass in das Genom eingegriffen werden muss.
Prof. Stich zu den weiteren Implikationen der Forschung: „Um beispielsweise die Ernährung angesichts des Klimawandels zu sichern, benötigen wir neue Variationen bekannter Nahrungspflanzen, die besser gegen neue Umweltbedingungen gewappnet sind und höhere Erträge bringen können. Hierfür legen wir im Exzellenzcluster CEPLAS die wissenschaftliche Grundlage. Mit unserer neuen Methode können wir nun systematisch hochrekombinogene Individuen züchten, um dann in einem Kreuzungsexperiment deutlich mehr Varianten zu erzeugen, um daraus vielversprechende Pflanzen zu selektieren.“
HHU
Originalpublikation:
Federico Casale, Delphine Van Inghelandt, Marius Weisweiler, Jinquan Li, and Benjamin Stich, Genomic prediction of the recombination rate variation in barley – A route to highly recombinogenic genotypes, Plant Biotechnology Journal (2021).
DOI: 10.1111/pbi.13746