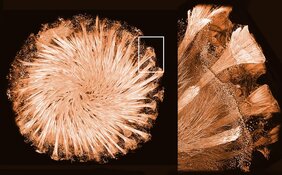
Glasschwämme haben - wie der Name vermuten lässt - ein Skelett auf Glasbasis, das aus einem Netz von Glasnadeln, Haken, Sternen und Kugeln besteht. Um eine solch einzigartige Architektur zu erreichen, müssen sie die Form von ungeordnetem Glas so manipulieren, dass hochgradig regelmäßige und symmetrische Elemente entstehen. Dünne kristalline Fasern aus einem Protein, dem so genannten Silikatein, befinden sich in Kanälen im Inneren dieser Glaselemente. Es ist bekannt, dass Silikatein-Kristalle für die Glassynthese in Schwämmen und für die Formgebung des Glasskeletts verantwortlich sind. Bisherige Versuche, die 3D-Struktur dieses Proteins zu bestimmen, zu beschreiben, wie es sich zu Kristallen zusammensetzt und wie diese das Glasskelett bilden, waren jedoch nicht erfolgreich. Vor allem, weil niemand in der Lage war, diese Kristalle im Labor zu reproduzieren.
Ein Team unter der Leitung von Dr. Igor Zlotnikov vom B CUBE - Center for Molecular Bioengineering an der TU Dresden versuchte einen ungewöhnlichen Ansatz. Statt Silikatein im Labor herzustellen und zu versuchen, Kristalle zur Untersuchung der Struktur zu erhalten, beschlossen die Forscherinnen und Forscher, die Glasnadeln aus einem Schwammskelett zu nehmen und die winzigen Kristalle zu analysieren, die bereits im Inneren existieren.
Zusammen mit Wissenschaftlerinnen und Wissenschaftlern des Dresden Center for Nanoanalysis (DCN) am Center for Advancing Electronics Dresden (cfaed) hat die Zlotnikov-Gruppe hochauflösende Transmissionselektronenmikroskopie (HRTEM) verwendet, um die in den Glasnadeln gepackten Silikatein-Kristalle genauer zu untersuchen. "Wir haben eine außergewöhnlich geordnete und gleichzeitig komplexe Struktur beobachtet. Bei der Analyse der Probe haben wir festgestellt, dass es sich um eine Mischung aus einem organischen und einem anorganischen Stoff handelt. Das bedeutet, dass sowohl Proteine als auch Glas eine hybride Überstruktur bilden, die irgendwie das Skelett von Schwämmen formt", erklärt Dr. Zlotnikov.
Eine traditionelle Methode zur Aufklärung der 3D-Struktur eines Proteins besteht darin, seinen Kristall einem Röntgenstrahl auszusetzen. Jeder Proteinkristall streut die Röntgenstrahlen auf unterschiedliche Weise und liefert so eine einzigartige Momentaufnahme seiner inneren Anordnung. Durch Drehen des Kristalls und Sammeln solcher Momentaufnahmen aus vielen Winkeln können die Forscher rechnergestützte Methoden zur Aufklärung der 3D-Proteinstruktur verwenden. Dieser Ansatz ist weit verbreitet und bildet die Grundlage der modernen Strukturbiologie. Er funktioniert gut für Kristalle mit einer Größe von mindestens 10 Mikrometern. Die Zlotnikov-Gruppe wollte jedoch Silikatein-Kristalle analysieren, die etwa 10 Mal kleiner waren. Wenn sie Röntgenstrahlen ausgesetzt waren, wurden sie fast sofort beschädigt, so dass es unmöglich war, einen vollständigen Datensatz aus mehreren Winkeln zu erfassen.
Daher griffen die Forschenden mit Unterstützung des Teams an der Synchrotron Lichtquelle Schweiz (SLS) am Paul Scherrer Institut zu einer neu aufkommenden Methode, der seriellen Kristallografie. "Man kombiniert dabei die Streuungsbilder von vielen Kristallen", erklärt Filip Leonarski, PSI-Strahllinienwissenschaftler, der an der Studie beteiligt war. "Mit der traditionellen Methode dreht man quasi einen Film. Mit der neuen Methode schiesst man viele Schnappschüsse, die man hinterher kombiniert, um die Struktur zu entschlüsseln." Jeder Schnappschuss wird an einer anderen Stelle dieser kleinen Kristalle gemacht oder sogar an einem anderen Kristall.
Insgesamt sammelten die Forschenden über 3500 einzelne Röntgenbeugungsmomentaufnahmen von 90 Glasnadeln in völlig zufälliger Orientierung. Mit modernsten Rechenmethoden konnten sie Ordnung in dem Chaos finden und die Daten zusammensetzen, um die erste vollständige 3D-Struktur des Silikateins aufzuklären.
"Bis zu dieser Studie wurde die Struktur von Silikatein aufgrund seiner Ähnlichkeit mit anderen Proteinen hypothetisch angenommen", sagt Dr. Zlotnikov. Mit Hilfe der neu gewonnenen 3D-Struktur des Silikateins konnten die Forscher seinen Aufbau und seine Funktion im Inneren des Glasskeletts des Schwamms verstehen und in Korrelation mit den komplexen HRTEM-Aufnahmen des Protein-Glas-Netzwerkes setzen.
"Wir haben detaillierte Informationen über eine funktionelle 3D-Protein-Glas-Protein-Superstruktur in einem lebenden Organismus geliefert. Tatsächlich handelt es sich bei dem, was wir beschreiben, um die erste bekannte natürlich vorkommende hybride Mineral-Protein-Kristallstruktur", schließt Dr. Zlotnikov.
TU Dresden
Originalpublikation:
Stefan Görlich, Abisheik John Samuel, Richard Johannes Best, Ronald Seidel, Jean Vacelet, Filip Karol Leonarski, Takashi Tomizaki, Bernd Rellinghaus, Darius Pohl and Igor Zlotnikov: Natural hybrid silica/protein superstructure at atomic resolution. PNAS (November 2020)