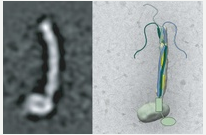
Mit hoher Genauigkeit konnten sie die Struktur des Komplexes aufklären und Einsichten in seine mechanische Funktionsweise erlangen. Die Ergebnisse sind in der Fachzeitschrift PNAS erschienen.
Im Inneren einer Zelle befinden sich zahlreiche unterschiedlich große, abgeschlossene Strukturen. Diese so genannten Organellen erfüllen spezifische Funktionen. Während Mitochondrien wie Kraftwerke Energie für die Zelle bereitstellen, entsprechen Peroxisomen am ehesten Kläranlagen. In ihnen arbeiten unterschiedliche Enzyme am Abbau schädlicher und an der Synthese nützlicher Stoffe. Dabei fällt das giftige Wasserstoffperoxid an, das umgehend von weiteren Enzymen im Peroxisom neutralisiert wird. Defekte der Peroxisomen oder ihrer Enzyme führen zu schweren Erkrankungen mit meist tödlichen Folgen.
Wie die meisten Organellen können auch Peroxisomen ihre Enzyme nicht selbst bilden. Stattdessen müssen die korrekten Enzyme auf die jeweiligen Organellen verteilt werden. Enzyme, die für das Peroxisom bestimmt sind, tragen eine von zwei möglichen Kennzeichnungen, die von Importrezeptoren im Zellinneren erkannt und von ihnen gebunden werden. Diese Importrezeptoren werden dann von einem Andock-Komplex zu Eintrittsporen in der Peroxisomenmembran geleitet, durch die die Enzyme aufgenommen werden. Ralf Erdmanns Team zeigte bereits in früheren Studien, dass die Enzyme durch unterschiedliche Poren eingeschleust werden, je nachdem, welche der beiden Signalsequenzen sie tragen. „Bisher sind jedoch nur wenige Details über diesen Prozess bekannt“, sagt Erdmann. Eine zentrale Rolle scheint der Andock-Komplex zu haben, der die Importrezeptoren an der peroxisomalen Membran einfängt. Nun ist es zum ersten Mal gelungen, die Architektur dieses Komplexes in nahezu atomarer Auflösung zu bestimmen. „Die enge Zusammenarbeit von Wissenschaftlerinnen und Wissenschaftlern mit Expertisen aus verschiedenen Fachgebieten war Schlüssel zum Erfolg“, betont Bettina Warscheid.
Wesentliche Bestandteile des Andock-Komplexes sind die beiden Proteine Pex14p und Pex17p. Tobias Hansen ist es in seiner Doktorarbeit an der Universität Bochum gelungen, einen hochreinen Komplex dieser Proteine zu isolieren. Pascal Lill am MPI in Dortmund entschlüsselte mit Hilfe der Kryo-Elektronenmikroskopie und -tomographie dessen Struktur: Sie besteht aus länglichen, parallel zueinander angeordneten Strängen, die gemeinsam wie ein Stab aus der Membran der Peroxisomen herausschauen. Daniel Wendscheck, Doktorand im Graduiertenkolleg 2202 „Transport über und in Membranen“ an der Universität Freiburg, zeigte mit nativer Massenspektrometrie und chemischer Quervernetzung, dass sich der Komplex aus drei Pex14p- und einem Pex17p-Protein zusammensetzt. Die gemeinsame Arbeit der Gruppen zeigt, dass flexible Bereiche der Pex14p-Proteine wie Tentakel in die Zelle ragen. Die Forschenden vermuten, dass die Tentakel die Importrezeptoren mit den Enzymen „angeln“, die für das Peroxisom bestimmt sind. „Dieser Komplex ist von essentieller Bedeutung für funktionstüchtige Peroxisomen“, sagt Christos Gatsogiannis. „Unser erstes Bild seiner Struktur und ein mechanistisches Modell seiner Funktion bilden eine wichtige Grundlage für weitere Untersuchungen der Peroxisomen und könnten auch zu einem besseren Verständnis peroxisomaler Krankheiten beitragen.“
Bettina Warscheid ist Leiterin der Abteilung Biochemie und Funktionelle Proteomforschung am Institut für Biologie II und Mitglied der Signalforschungszentren BIOSS und CIBSS an der Universität Freiburg. Ralf Erdmann ist Leiter der Abteilung Systembiochemie am Institut für Biochemie und Pathobiochemie der Universität Bochum. Christos Gatsogiannis war bis vor Kurzem Projektgruppenleiter am Max-Planck-Institut für molekulare Physiologie in Dortmund und hat im September einen Ruf als Professor für Elektronen-Tomographie nanostrukturierter Systeme an das Neue „Center for Nanoscience“ der Westfälischen Wilhelms-Universität Münster angenommen. Die Zusammenarbeit erfolgte im Rahmen der von der Deutschen Forschungsgemeinschaft geförderten Forschungsgruppe „Struktur und Funktion des peroxisomalen Translokons“.
Albert-Ludwigs-Universität Freiburg
Originalpublikation:
Lilla, P./Hansen, T./Wendscheck, D./Klink, B.U./Jeziorek, T./Vismpas, D./Miehlinga J./Bender, J./Schummer, A./Drepper, F./Girzalsky, W./Warscheid, B./Erdmann, R./Gatsogiannisa, C. (2020): Towards the molecular architecture of the peroxisomal receptor docking complex. In: Proceedings of the National Academy of Sciences of the United States of America (PNAS).