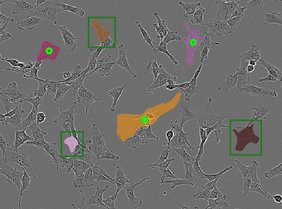
Es ist bereits möglich, biologische Strukturen automatisch zu segmentieren. Das Problem bei bisherigen Methoden: Die Anwendung funktioniert nur unter bestimmten Bedingungen und die Anpassung an neue Bedingungen ist sehr aufwendig. Um die bestehende Software zu verbessern, haben die Forschenden das Modell mit einem Mikroskopie-Datensatz von über 17.000 Mikroskopiebildern mit über zwei Millionen von Hand markierten Strukturen neu trainiert. Dadurch verbesserte sich die Leistung des Modells bei der Segmentierung von Zellbestandteilen drastisch. Das Team entwickelte daraufhin die Software μSAM, die es Forschenden und medizinischem Fachpersonal ermöglicht, Bilder zu analysieren, ohne vorher Strukturen manuell zeichnen oder ein spezielles KI-Modell trainieren zu müssen.
„Zellen zu analysieren zählt zu den anspruchsvollsten Aufgaben der Mikroskopie. Der Prozess ist sowohl für die biologische Grundlagenforschung als auch für die medizinische Diagnostik essenziell“, erklärt Prof. Dr. Constantin Pape vom Institut für Informatik der Universität Göttingen. „Meine Forschungsgruppe ist darauf spezialisiert, Methoden zur Automatisierung solcher Aufgaben zu entwickeln. Deshalb fragen uns Forschende oft nach Hilfe. Vor der Entwicklung von Segment Anything for Microscopy mussten wir sie bitten, viele Strukturen zunächst von Hand zu markieren – eine schwierige und zeitaufwändige Aufgabe. μSAM hat dies geändert: Aufgaben, die früher wochenlange, mühsame Handarbeit erforderten, können in wenigen Stunden automatisiert werden. Danach kann das Modell weiter verbessert werden. Das eröffnet viele neue Anwendungsbereiche: Wir haben es bereits in zahlreichen Projekten eingesetzt, die von der grundlegenden Zellbiologie bis hin zur Entwicklung von Methoden für Behandlungsempfehlungen bei Krebstherapien reichen“, so Pape.
Universität Göttingen
Originalpublikation:
Archit, A., Freckmann, L., Nair, S. et al. Segment Anything for Microscopy. Nat Methods (2025). doi.org/10.1038/s41592-024-02580-4