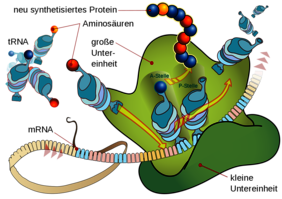
Die Regulation der Genaktivität ist ein elementarer Prozess des Lebens. Dabei wird die genetische Information, die in Form von DNA im Zellkern gespeichert ist, in das Botenmolekül mRNA umgeschrieben. Dieses wird dann vom Ribosom abgelesen und in ein Protein übersetzt. Damit die Zelle richtig funktionieren kann, muss dieser Prozess auf mehreren Ebenen streng kontrolliert werden. Eine entscheidende Komponente ist dabei auch die Frage, wann und wie die Boten-RNA am Ende wieder abgebaut wird. Ein internationales Team um Professor Roland Beckmann vom Genzentrum der LMU hat in Kooperation mit Jeff Coller aus den USA und Toshifumi Inada aus Japan erstmals die Struktur eines am Abbau beteiligten Komplexes aufgeklärt und die molekularen Mechanismen aufgedeckt. Wie die Wissenschaftler im renommierten Fachmagazin Science berichten, können sie so erstmals erklären, warum und auf welche Weise der mRNA-Abbau mit der Effizienz der Proteinproduktion zusammenhängt.
„Statistische Daten hatten bereits gezeigt, dass der mRNA-Abbau an die Geschwindigkeit des Ribosoms bei der Proteinproduktion gekoppelt ist“, sagt Robert Buschauer, Doktorand in Beckmanns Team und Erstautor des Papers. „Wie dies auf molekularer Ebene funktioniert, war allerdings völlig unbekannt.“
Die Effizienz des Ribosoms hängt davon ab, wie gut es den genetischen Code der mRNA lesen kann: Der Code legt fest, welche Aminosäure als nächstes in das wachsende Protein eingebaut wird. Diese Aminosäure wird von sogenannten tRNA-Molekülen zum Ribosom gebracht. Für fast alle Aminosäuren existieren allerdings mehrere Codes, die unterschiedlich gut vom Ribosom gelesen werden können. Bei schlecht lesbaren Codes braucht es länger, um die passende Aminosäure zu finden, sodass die tRNA-Bindungsstelle länger unbesetzt bleibt. Nun haben die Wissenschaftler die Verbindung zwischen diesem Effekt und dem mRNA-Abbau gefunden: „Wir konnten mithilfe von Kryoelektronenmikroskopie zeigen, dass ein Schlüsselkomplex des mRNA-Abbaus nur an das Ribosom binden kann, wenn keine tRNA am Ribosom sitzt.“ Die Wahrscheinlichkeit, dass es einen Abbauprozess gibt, steigt also mit dem Anteil schlecht lesbarer Codes.
„Diese von uns gezeigte Interaktion ist tatsächlich entscheidend für die Kopplung von mRNA Abbau und der Effizienz des Ribosoms“, sagt Beckmann. Der mRNA-Abbau ist ein grundlegender Prozess, dessen zentrale Elemente in Organismen von der Hefe bis zum Menschen nahezu identisch funktionieren. Treten dabei Fehler auf, können unter anderem neurodegenerative Erkrankungen oder Krebs entstehen. Ein besseres Verständnis der Mechanismen ist daher auch wichtig für die Entwicklung neuer therapeutischer Ansätze.
(Ludwig-Maximilians-Universität München)
Originalpublikation:
Robert Buschauer, Yoshitaka Matsuo, Takato Sugiyama, Ying-Hsin Chen, Najwa Alhusaini, Thomas Sweet, Ken Ikeuchi, Jingdong Cheng, Yasuko Matsuki, Risa Nobuta, Andrea Gilmozzi, Otto Berninghausen, Petr Tesina, Thomas Becker, Jeff Coller, Toshifumi Inada and Roland Beckmann :
The Ccr4-Not complex monitors the translating ribosome for codon optimality, Science 2020